第2章 daisy-toolsの使い方
2.1 主に使用するツール類
daisy-toolsは様々なツールで構成されています。主に使用するツールは表2.1の通りです。なお、これらのファイルはdaisy-toolsのGitHubリポジトリのReleasesページからダウンロードできます(後述)。
表2.1: daisy-toolsの基本的なツール
| ツール名 | 機能/役割 |
|---|---|
| dsy-sysenv | 周期的な細胞の実行を行う。 daisy-toolsのシステムを担うメインの実行バイナリ。 |
| dsy-cell2elf | daisy-toolsの細胞形式のバイナリをELFバイナリへ変換する。 |
| dsy-dump-cell | 細胞形式バイナリの内容を独自のリスト形式 あるいはJSON形式でダンプする。 |
| dsy-name | 生成される細胞バイナリ・化合物バイナリのファイルの 名前付けを行うスクリプト。(ユーザー定義) |
| dsy-eval | 各周期でdsy-sysenvから呼び出され、指定された細胞を評価し 適応度を返すスクリプト。(ユーザー定義) |
dsy-sysenvはカレントディレクトリにリスト2.1の構成で配置された細胞ファイル・化合物ファイルを認識します。
.
|-- bin
| |-- "dsy-"で始まる各ツールを配置
| |-- dsy-sysenv
| |-- dsy-cell2elf
| |-- dsy-dump-cell
| |-- dsy-name <- ユーザー定義スクリプト
| |-- dsy-eval <- ユーザー定義スクリプト
| `-- ...etc
|-- cell
| `-- 細胞ファイルを配置
|-- code
| `-- コード化合物ファイルを配置
|-- data
| `-- データ化合物ファイルを配置(現バージョンでは未使用)
`-- samples
`-- 細胞ファイル・コード化合物ファイルを生成するサンプルツール群
リスト2.1のディレクトリ構成のカレントディレクトリ上でdsy-sysenvを実行すると、存在する全ての細胞ファイルに対して「代謝/運動」・「成長」・「増殖」・「死」といった生物の振る舞いを繰り返し実行します。
dsy-nameとdsy-evalは、dsy-sysenvから必要に応じて呼び出されるユーザー定義のスクリプトで、dsy-sysenv実行前に予め用意しておく必要があります。
以降ではそれぞれのファイルについて説明します。
dsy-nameについて
dsy-sysenvが新たに細胞ファイルやコード化合物ファイルを生成する際に呼び出すスクリプトで、それらのファイル名を決めるために使用します。
- 第1引数に"cell"あるいは"code"を指定して呼び出される
- 名付ける対象が細胞ファイル/コード化合物ファイルのどちらなのかを指定
dsy-sysenvは標準出力に出されたものをファイル名として扱う- 指定されたファイル名が既に存在する場合、
dsy-sysenvはdsy-nameを繰り返し呼び出す- 現時点(2020年2月現在)では、存在しないファイル名が返されるまでリトライし続ける実装
生成される細胞や化合物には固有の名前が付けられると面白いかなと思い、ファイル名の名付けの部分は別スクリプトへ切り出しました。
ただ、動作上、ファイル名は何でも良いので、例えばリスト2.2の様にランダムな文字列を返すようにしてしまっても構いません。
#!/bin/bash echo $RANDOM | md5sum | cut -c-10
なお、リスト2.2はsamples/dsy-nameに用意してあります。これをそのまま使う場合は、binディレクトリにdsy-nameという名前でこのファイルのシンボリックリンクを作成すれば良いです。(もちろんWindows等の環境で実行する場合はファイルコピーでも構いません。)
dsy-evalについて
dsy-evalは、各周期で各細胞の適応度を決めるために細胞毎に呼び出されるスクリプトです。ユーザーはこのファイルで細胞をどのように評価するかを定義します。
- 第1引数に対象の細胞ファイル名を指定して呼び出される
- "cell"から始まるパス指定ではなく、細胞ファイル名のみの指定
dsy-sysenvはdsy-evalの終了ステータスの値を適応度として扱う- 適応度の範囲は0から100
- 100の適応度が返された場合、
dsy-sysenvは目的のバイナリが生成できたとして実行終了する - 0から100以外の終了ステータスを返された場合、
dsy-sysenvはエラー終了する
評価の流れを図示すると図2.1の通りです。
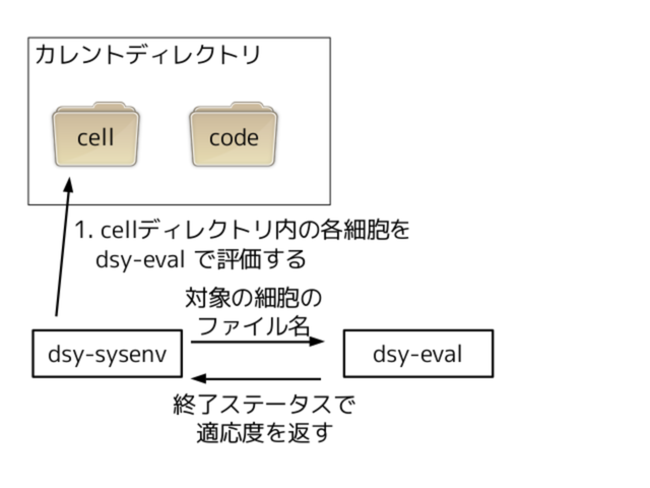
図2.1: dsy-evalを使用した評価の流れ
例えば「細胞がエラーなく終了できるか(終了ステータスが0であるか)」のみを評価するようなものはシェルスクリプトでリスト2.3のように記述します。
#!/bin/sh
set -uex
if [ $# -ne 1 ]; then
echo "Usage: $0 CELL_FILE" 1>&2
exit 200
fi
cell_file=$1
bin/dsy-cell2elf $cell_file ${cell_file}.elf
chmod +x ${cell_file}.elf
./${cell_file}.elf && fitness=100 || fitness=50
rm ${cell_file}.elf
exit $fitness
dsy-cell2elfは第1引数で指定された細胞ファイルをELFバイナリへ変換し、第2引数で指定されたパスへ保存します。
リスト2.3では、dsy-cell2elfで変換したELFバイナリを試しに実行し、正常終了したら適応度として終了ステータス100を、異常終了したら適応度として終了ステータス50を返すようにしています。
適応度は、細胞が化合物を取り込める確率として使用されるものなので、0を返してしまうと、その細胞は今後一切化合物を取り込めなくなり、細胞分裂も行えなくなってしまう点にご注意ください。また、0で無くとも、あまりにも低い適応度ばかりを与えると適応度100の細胞が生まれる前に環境の細胞が全滅してしまう点も注意が必要です。経験的には最低適応度は50くらいにしておくと良い感じです。
リスト2.3のスクリプトは、samples/dsy-eval-exit_0にあります。これをそのまま使う場合は、このファイルのシンボリックリンクかコピーをbinディレクトリにdsy-evalという名前で作成してください。
本書でdsy-evalとして使用する評価スクリプトは全てsamplesディレクトリに置いてあります。
2.2 使用例: 簡単な評価スクリプトで試す
ここでは、使い方の紹介として、前節の評価スクリプトを使用して、次章でも実験結果として紹介する「正常終了するだけのELFバイナリ生成」へ向けた環境を作ってみます。
ツールをダウンロード
daisy-toolsリポジトリのReleasesページでビルド済みバイナリをZIPアーカイブで配布しています。
- daisy-toolsリポジトリのReleasesページ
ダウンロードしたらお好きな場所へ展開してください。dsy-workというディレクトリができあがります。このディレクトリ名は変更しても構いません。
なお、現行のビルド済みバイナリの動作環境は表2.2の通りです。
表2.2: 動作環境
| ライブラリ/ソフトウェア | バージョン |
|---|---|
| Linux | 特になし。ELFが実行できればOK。 |
| GNU C ライブラリ(libc6) | 2.24 |
| bash | 特になし。bashのシェルスクリプトが実行できればOK。 |
標準的なLinux環境であればディストリビューション問わず動作すると思います*1。
[*1] 筆者が使用しているのはDebian GNU/Linux 9 (stretch)です。
ただ、必須要件ではないのですが、ログのほとんどはsyslogに出力しますので、syslogが使えない環境(WSL等)ではsyslog出力のログは見ることができません。
細胞ファイルを配置
進化のベースとなる最初の状態の細胞ファイルをcellディレクトリへ配置します。
本書で進化のベースとして使用する細胞ファイルは以下のいずれかです。
cell/initialret命令のみの細胞- 自身を構成する命令には突然変異不可フラグ設定済み
samples/create-init-cellで生成可能
cell/exit- 終了ステータス0で終了するための機械語命令群と
ret命令の細胞 - 自身を構成する命令には突然変異不可フラグ設定済み
samples/create-exit-cellで生成可能
- 終了ステータス0で終了するための機械語命令群と
それぞれのツールを実行すると、cellディレクトリにそれぞれのファイル名で細胞ファイルを生成します。なお、daisy-toolsでは今の所、ファイル名で何らかの判断は行っていないので、ファイル名は変更しても構いません。
例えば、initialの細胞ファイルをcellディレクトリへ配置する場合は、以下のようにsamples/create-init-cellを実行します。
$ samples/create-init-cell $ ls cell/ initial
コード化合物ファイルを配置
コード化合物ファイルは、細胞が成長や増殖する際に使用するコード化合物に当たるファイルです。表2.3の構成の16バイトのファイルです。
表2.3: コード化合物ファイルの構成
| オフセット | 内容 | 長さ[バイト] |
|---|---|---|
| 0 | 命令の長さ[バイト] | 8 |
| 8 | 機械語命令 | 8 |
なお、dsy-sysenvではコード化合物ファイル1つを1命令として扱います。そのため、分割してほしくない複数の命令がある場合、1つのコード化合物ファイルに並べておけば、1命令として扱われます。
本書で生成する「正常終了するだけの実行バイナリ」と「文字'A'を出力する実行バイナリ」については、生成するために必要なコード化合物ファイルは、それぞれ以下のサンプルツールで生成できます。
- 正常終了するだけの実行バイナリ
- 必要なコード化合物ファイルは
samples/create-exit-codesで生成できる
- 必要なコード化合物ファイルは
- 文字'A'を出力する実行バイナリ
- 必要なコード化合物ファイルは
samples/create-puta-codesで生成できる
- 必要なコード化合物ファイルは
それぞれのツールは共に、実行時に「各命令で何個ずつコード化合物ファイルを生成するか」をコマンドライン引数で指定します。そして実行すると、指定した個数ずつ、各命令のコード化合物ファイルがcodeディレクトリへ生成されます。
例えば、「正常終了するだけの実行バイナリ」に必要なコード化合物ファイルを生成するには以下の通りです。(各命令100個ずつ生成)
$ samples/create-exit-codes 100
+ '[' 1 -ne 1 ']'
+ num_codes=100
+ mkdir -p code
++ seq -w 100
+ for i in $(seq -w ${num_codes})
...
$ ls -1 code
ret_001
..
ret_100
syscall_60_001
...
syscall_60_100
xor_rdi_rdi_001
...
xor_rdi_rdi_100
このサンプルでは各コード化合物ファイルのファイル名を各命令になぞらえて付けていますが、細胞ファイル同様にファイル名は何でも構いません。
なお、実行ログからも分かる通り、samples/create-exit-codesのサンプルツールはシェルスクリプトです。簡単なコマンドでコード化合物ファイルを生成する際に参考にしてみてください。
ユーザー定義スクリプトを配置
binディレクトリにdsy-nameとdsy-evalのユーザー定義スクリプトを配置してください。
前述の通り、dsy-nameはsamples/dsy-nameのシンボリックリンクかコピーで構いません。
$ ln -s ../samples/dsy-name bin/dsy-name $ ls -l bin/dsy-name lrwxrwxrwx 1 yohgami yohgami 19 2月 12 14:59 bin/dsy-name -> ../samples/dsy-name
dsy-evalは生成したいバイナリによって変える必要があります。前節で説明した評価スクリプトで試す場合は以下のようにシンボリックリンクを作成します。
$ ln -s ../samples/dsy-eval-exit_0 bin/dsy-eval $ ls -l bin/dsy-eval lrwxrwxrwx 1 yohgami yohgami 26 2月 12 15:02 bin/dsy-eval -> ../samples/dsy-eval-exit_0
dsy-sysenvを実行
以上で準備は完了です、dsy-sysenvを実行すると環境の動作が始まり、細胞の生命活動が始まります。
$ bin/dsy-sysenv
dsy-sysenvを実行するとrunningというファイルがカレントディレクトリに作成されます。中断したい場合はこのファイルを削除してください。すると、その周期の全ての動作を終えたところでdsy-sysenvは終了します。
$ ls running running $ rm running
dsy-sysenvは現在の環境の状態をcellとcodeディレクトリに保存しています。再開したい場合は単にdsy-sysenvをもう一度実行すれば良いです。
そして、適応度が100の細胞が生まれると、dsy-sysenvはその細胞ファイルをカレントディレクトリにout.cellという名前で保存し、その周期を実行し終えた後、プログラム自体を終了します。