図3.1: 部分交叉について
本書で扱う遺伝的アルゴリズムの流れを説明します。一般的な定義と大きく違うところは無いと思いますが、筆者は遺伝的アルゴリズムに詳しいわけでは無いので、中には本書独自のところもあるかと思います。
本書ではMBRを遺伝的アルゴリズムにおける一つ一つの個体として扱い、遺伝的アルゴリズムを適用してみます。この場合、MBRを構成するバイナリ列が「遺伝子*1」となります。
[*1] 「遺伝子」という言葉は、本来、DNAの中でタンパク質の作り方を記録している部分を指しますが、本書では(というより遺伝的アルゴリズムの世界では?)「遺伝に関する情報」や「各個体を形作るデータ列」といった意味で「遺伝子」という言葉を使っています。
遺伝的アルゴリズムは大きく以下の流れで行います。
それぞれについて簡単に説明します。(詳細は後述します。)
「1. 初期個体群生成」は、単にMBRのバイナリ列を個体数分用意するだけです。
「2. 評価」で前章で作ったMBRテスターを使います。次のステップのために各個体に対する評価結果を保存しておきます。
「3. フィードバック」では評価結果を個体群へフィードバックします。まず評価結果の悪かった個体を淘汰し、優秀な個体はそのままコピーして増やします。そして、上位何割かで遺伝子を交叉させて子を作り、また何割かで遺伝子の突然変異を起こします。
以降、2と3を繰り返すことで目的のMBRへ近づけていきます。なお、2と3を行う1サイクルを「世代」と呼ぶことにします。「評価とフィードバックが終わると現在の世代が終わり、次のループから次の世代が始まる」というイメージです。
本書では、完全に筆者の趣味で、シェルスクリプトで実装しています。以下のリポジトリで公開しています。
ただ、シェルスクリプトは一般的にあまり読みやすいものではないです。そこで、ここでは先述した大きく3つの処理それぞれについて、どのように実装したのかを説明していくことにします。
そんなに大したものではないので、もし興味があれば自身のお好きな言語で実装してみることをおすすめします。
リポジトリ内の"ge_mbr.sh"に全て書かれています。
先述した3つの処理は関数化していますので、それらの説明は後でするとして、ここでは全体の処理をざっと説明します。「実装の詳細は置いておいて、ひとまず遊んでみたい」という場合もこの節さえ読んでもらえれば大丈夫です。
まず、グローバル変数を定義します(リスト3.1)。
リスト3.1: グローバル変数定義
#!/bin/bash # set -eux # POPULATION_SIZEは、 # - 2で2回割っても余りが出ない # - 5で1回割っても余りが出ない # を共に満たす値であること # すなわち、20の倍数 POPULATION_SIZE=20 GENE_LEN=446 MBR_TESTER=./mbr_tester WORK_DIR=$(date '+%Y%m%d%H%M%S') # ・・・以降、関数定義が続く・・・
"POPULATION_SIZE"は個体数です。後の処理の都合上、コメントの通り20の倍数である必要があります。
GENE_LENは遺伝子の長さ(バイト数)です。MBR512バイトは、「イニシャルプログラムローダ(IPL)」(446バイト)と「パーティションテーブル」(64バイト)と「ブートシグネチャ」(2バイト)で構成されています。遺伝的アルゴリズムで求めたいのはBIOSによってロードされる実行バイナリ部分であるIPLなので、446を遺伝子の長さとしています。とはいえ、MBRテスターで実行する際は512バイト揃っていなければならないため、パーティションテーブルとブートシグネチャのバイナリは"mbr_partition_tbl_boot_sig.dat"というバイナリでリポジトリ内に予め用意してあり、IPLのバイナリに結合して512バイトのMBRを作ります。
WORK_DIRは作業ディレクトリです。この中に全固体の全世代のデータが保存されていきます。
関数定義部分は飛ばして、次はコマンドライン引数チェックです(リスト3.2)。
リスト3.2: コマンドライン引数チェック
if [ $# -ge 1 ]; then
WORK_DIR=$1
echo "Use WORK_DIR=${WORK_DIR}"
else
echo "Initialization"
initialization
fi
if [ $# -ge 2 ]; then
age=$2
echo "It will start in the generation $age."
else
age=0
fi
ge_mbr.shは途中再開のための引数を最大2つとります。
$ ./ge_mbr.sh [作業ディレクトリ] [世代番号]
それぞれの引数が指定されれば、指定されたディレクトリで指定された世代番号から再開します。
指定されなかった場合は、作業ディレクトリは先述したディレクトリで、世代番号0から始めます。
次に、メイン処理が続きます(リスト3.3)。
リスト3.3: メイン処理
touch enable_ge
while [ -f enable_ge ]; do
echo "GE: age=$age"
echo '-------------------------------------------'
evaluation
feedback
mv ${WORK_DIR}/{now,$age}
mv ${WORK_DIR}/{next,now}
mkdir ${WORK_DIR}/next
age=$((age + 1))
echo
echo
done
「評価(evaluation関数)」と「フィードバック(feedack関数)」をwhileで繰り返しています。
whileの直前でtouchにより空ファイルを生成し、whileのループ条件にもなっている"enable_ge"ファイルは、ge_mbr.shスクリプトを中断させるためのファイルです。evaluation関数やfeedback関数では作業ディレクトリ内で適宜ファイルを置いたり移動させたりしながら処理を行います。これらの作業中にCtrl+C等で終了させてしまうと作業ディレクトリ内が中途半端な状態になり、再開させることが難しくなります。そのため、enable_geファイルを用意し、このファイルを消すことで現世代の処理を終えたらwhileループを抜けることができるようにしています。
評価とフィードバックが終わったところで行っているのは作業ディレクトリ内のディレクトリ名の更新です。
ここで、作業ディレクトリ内の構成について説明します。ある時点の作業ディレクトリ内はリスト3.4のようになっています。
リスト3.4: ある時点の作業ディレクトリの状態
20180912144115/ ├─ 0/ │ ├─ ch_0.dat │ ├─ ch_1.dat │ ├─ ch_2.dat │ ├─ ... │ └─ evaluation_value_list.csv ├─ 1/ │ ├─ ch_0.dat │ ├─ ch_1.dat │ ├─ ch_2.dat │ ├─ ... │ └─ evaluation_value_list.csv ├─ now/ │ ├─ ch_0.dat │ ├─ ch_1.dat │ ├─ ch_2.dat │ ├─ ... │ └─ evaluation_value_list.csv └─ next/
"ch_0.dat"や"ch_1.dat"というファイルそれぞれが512バイトのMBRです。"now"ディレクトリは現在の世代です。evaluation関数での評価は"now"ディレクトリに対して行います。また、"next"ディレクトリは次の世代を示すディレクトリで、feedback関数で次の世代の個体群を作るために使います。"0"や"1"等の数字のディレクトリは過去の世代のディレクトリです。
なお、各ディレクトリに存在する"evaluation_value_list.csv"はその世代の全固体のファイル名と評価によって得られた評価値のCSVです。evaluation関数内で作成します。
リスト3.3のディレクトリ名更新処理に説明を戻すと、フィードバックまでを終えるとnextディレクトリには次の世代の個体群が揃った状態になっているので、現世代の"now"ディレクトリを現在の世代番号($age)へリネームし、次世代の"next"ディレクトリを現世代を示す"now"ディレクトリへリネームしています。すると"next"ディレクトリが無くなるのでmkdirで作成し、世代番号をインクリメントします。
最後に再開させる際のコマンドを出力します(リスト3.5)。
リスト3.5: スクリプト終了処理
echo '-------------------------------------------'
echo "Stopped at $(date '+%Y-%m-%d %H:%M:%S')"
echo 'Continue Command'
echo "$0 ${WORK_DIR} $age"
echo '-------------------------------------------'
出力例はリスト3.6の通りです。
リスト3.6: 終了処理の出力例
------------------------------------------- Stopped at 2018-09-14 15:17:15 Continue Command ./ge_mbr.sh 20180912144115 821 -------------------------------------------
この場合、再開させる際は"./ge_mbr.sh 20180912144115 821"というコマンドで再開できます。
ここまでで、スクリプト全体を俯瞰した解説は終わりです。このスクリプトを単に道具として使ってみたい場合はここらへんまでで、遊んでみてください。
先述した「初期個体生成」・「評価」・「フィードバック」の実装に興味がある場合は、次節からの説明をご覧ください。
初期個体群生成を行うinitialization関数はリスト3.7の通りです。
リスト3.7: 初期個体群生成
initialization() {
local i
echo 'initialization'
echo '-------------------------------------------'
echo "WORK_DIR=${WORK_DIR}"
mkdir -p ${WORK_DIR}/{now,next}
for i in $(seq 0 $((POPULATION_SIZE - 1))); do
echo "${WORK_DIR}/now/ch_$i.dat"
generate "${WORK_DIR}/now/ch_$i.dat"
done
echo
echo
}
作業ディレクトリ内にnowとnextのディレクトリを作成し、generate関数を呼び出して個体数分の"ch_番号.dat"ファイルをnowディレクトリに生成します。
generate関数の実装はリスト3.8の通りで、引数で受け取ったファイル名のMBRのIPL部分をランダムで生成するだけです。
リスト3.8: 各個体の生成
generate() {
local file
file=$1
dd if=/dev/urandom of=$file bs=1 count=$GENE_LEN
cat mbr_partition_tbl_boot_sig.dat >> $file
}
次に、現世代の評価を行うevaluation関数の実装はリスト3.9の通りです。
リスト3.9: 評価
evaluation() {
local i
local ch
echo 'evaluation'
echo '-------------------------------------------'
rm -f tmp.csv
for i in $(seq 0 $((POPULATION_SIZE - 1))); do
echo "i=$i"
ch=${WORK_DIR}/now/ch_$i.dat
echo ">>>>> $ch"
cp $ch floppy.img
cp $ch hdd.img
$MBR_TESTER
exit_code=$?
case $exit_code in
0)
evaluation_value=100
echo "$ch has reached the answer!"
rm -f enable_ge
;;
1)
evaluation_value=1
echo "$ch is an ordinary child."
;;
*)
evaluation_value=0
echo "$ch is dead."
;;
esac
echo "ch_$i.dat,${evaluation_value}" >> tmp.csv
echo "evaluation_value=${evaluation_value}"
echo
echo
done
# 第2列で降順にソート
sort -n -r -k 2 -t ',' tmp.csv > ${WORK_DIR}/now/evaluation_value_list.csv
rm -f floppy.img hdd.img tmp.csv
echo
echo
}
少し行数が多いですが、やっていることは大したことではありません。
個体数の分だけforループを回し、各個体のMBRファイルでMBRテスターを実行しています。
実行後、caseで終了ステータスで条件分岐しています。
0はテストをpassし、EXIT_SUCCESSでMBRテスターが終了した場合なので、評価値(evaluation_value変数)に最高の100を設定し、答えに到達した旨をechoしています。その後、enable_geを削除し現世代でスクリプトを終了させるようにしています。
1はテストがfailし、EXIT_FAILUREでMBRテスターが終了した場合なので、評価値には1を設定し、この個体は平凡であった旨を出力します。
それ以外は、この個体のMBRによってMBRテスターが異常終了した場合なので、評価値には最低の0を設定し、その個体は死んだ旨を出力します(feedback関数で淘汰されます)。
個体数分のforループを終えると、全個体の評価値の降順でソートしたCSV(evaluation_value_list.csv)を生成し、evaluation関数は終わりです。このCSVを次のfeedbackn関数で使います。
最後にfeedback関数の実装ですが、結構量があるので部分毎に分けて説明します。
feedback関数では、まず、feedback関数内で使用する定数を定義します(リスト3.10)。
リスト3.10: フィードバック(定数定義)
feedback() {
local i
echo 'feedback'
echo '-------------------------------------------'
candidates_num=$((POPULATION_SIZE / 2))
mutation_num=$((POPULATION_SIZE / 5))
top_num=$((POPULATION_SIZE - (candidates_num + mutation_num)))
"candidates_num"は交叉を行う候補となる個体の数です。全個体数の上位50%で交叉を行い、同数の子供を次世代へ引き継ぎます。
"mutation_num"は突然変異させる個体の数です。交叉を行う候補とは別で選定し、突然変異させた後、次世代へ引き継ぎます。
"top_num"は何も変化を与えずにそのまま引継ぐ個体の数です。交叉をさせる50%と突然変異させる20%を除いた残り30%をそのまま次世代へ引き継ぎます。30%の個体は評価値上位から選定します。
次に、evaluation関数で評価値が0になった個体を淘汰します(リスト3.11)。
リスト3.11: フィードバック(淘汰)
# 淘汰
# evaluation_value=0の個体を一番良い個体で置き換える
echo ">>>>>>> To be culled"
top_ch=$(awk -F',' '{print $1;exit}' \
${WORK_DIR}/now/evaluation_value_list.csv)
echo "top_ch:${WORK_DIR}/now/${top_ch}"
for i in $(awk -F',' '$2==0{print $1}' \
${WORK_DIR}/now/evaluation_value_list.csv); do
mkdir -p ${WORK_DIR}/${age}_culled
mv ${WORK_DIR}/now/${i} ${WORK_DIR}/${age}_culled/
echo "save $i to ${WORK_DIR}/${age}_culled/"
cp ${WORK_DIR}/now/${top_ch} ${WORK_DIR}/now/$i
echo "replace ${WORK_DIR}/now/$i with ${WORK_DIR}/now/${top_ch}"
done
echo
淘汰は、評価値が0であった個体について、nowディレクトリ内の当該個体を、一番評価値が高かった個体で置き換えることで行っています。
これだけだと淘汰されたMBRは上書きされて失われてしまうので、淘汰が発生する際は"<世代番号>_culled"というディレクトリを作成し、そこへ淘汰されるMBRを事前にバックアップしています。
また、nowディレクトリ内のファイルだけ上書きして、CSVには反映させていません。これは交叉の際に候補となるようにしており、詳しくは後述します。
次に、評価値上位から全数の30%の個体を次世代へ引き継ぎます(リスト3.12)。
リスト3.12: フィードバック(上位30%を引継ぐ)
# (個体数 - (残した子孫の数 + 突然変異数))分の個体を、上位から順にnextへコピー
echo ">>>>>>> Copy Top ${top_num}"
for i in $(seq ${top_num}); do
line=$(sed -n ${i}p ${WORK_DIR}/now/evaluation_value_list.csv)
echo "line:$line"
cp ${WORK_DIR}/{now,next}/$(echo $line | cut -d',' -f1)
echo "save_to_next:${WORK_DIR}/now/$(echo $line | cut -d',' -f1)"
done
echo
top_numの数だけループを回し、評価値上位から順にnextディレクトリへコピーしています。
続いて、全数の50%で交叉を行います(リスト3.13)。
リスト3.13: フィードバック(交叉)
# 上位50%の個体を候補に、同数の個体をnextへ追加
# (一様交叉を使用)
echo '>>>>>>> Crossover'
max_cross_times=$((candidates_num / 2))
for cross_times in $(seq 0 $((max_cross_times - 1))); do
echo "cross_times=${cross_times}"
chA_idx=$(((RANDOM % candidates_num) + 1))
chB_idx=${chA_idx}
while [ ${chB_idx} -eq ${chA_idx} ]; do
chB_idx=$(((RANDOM % candidates_num) + 1))
done
echo "chA_idx=${chA_idx} , chB_idx=${chB_idx}"
chA=${WORK_DIR}/now/$(sed -n ${chA_idx}p \
${WORK_DIR}/now/evaluation_value_list.csv \
| cut -d',' -f1)
chB=${WORK_DIR}/now/$(sed -n ${chB_idx}p \
${WORK_DIR}/now/evaluation_value_list.csv \
| cut -d',' -f1)
echo "chA=$chA"
echo "chB=$chB"
partial_crossover $chA $chB \
child$((2 * cross_times)).dat \
child$(((2 * cross_times) + 1)).dat
echo
done
cross_idx=0
for i in $(seq 0 $((POPULATION_SIZE - 1))); do
[ ${cross_idx} -ge ${candidates_num} ] && break
if [ ! -f ${WORK_DIR}/next/ch_$i.dat ]; then
mv child${cross_idx}.dat ${WORK_DIR}/next/ch_$i.dat
echo "mv child${cross_idx}.dat ${WORK_DIR}/next/ch_$i.dat"
cross_idx=$((cross_idx + 1))
fi
done
echo
まず、1つ目のループでは交叉の回数分(候補数の半分)ループを回し、交叉対象の選択と交叉を行っています。変数chAとchBを設定するところまでが交叉対象の選択です。交叉対象は評価結果の上位50%の中から任意の2つを選択することで決めます。そして、交叉はpartial_crossoverという関数で行っています。
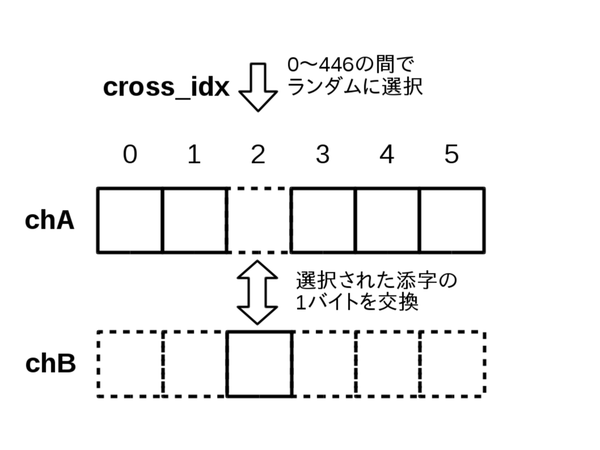
partial_crossover関数(部分交叉)はコードを見せるよりも図で示すと一目瞭然なので、図3.1に示します。単にランダムに選んだ1バイトを互いに入れ替えるだけです。

図3.1: 部分交叉について
なお、現在の実装では一度交叉を行った個体が再度選択される可能性があります。子供を残す個体も居れば、残さない個体も居る、という方が自然かなと思い、このようにしています*2。
[*2] と言いつつ、単に実装をサボっただけでもあります。
そして、2つ目のループでは1つ目のループで作った子供たちに番号を割り当て、nextディレクトリに"ch_番号.dat"というファイル名で保存しています。
最後に、残る20%の個体を突然変異させます(リスト3.14)。
リスト3.14: フィードバック(突然変異)
# 個体数の20%を突然変異させ、nextへ追加
echo '>>>>>>> Mutation'
for i in $(seq 0 $((POPULATION_SIZE - 1))); do
if [ ! -f ${WORK_DIR}/next/ch_$i.dat ]; then
generate ${WORK_DIR}/next/ch_$i.dat
echo "mutated:${WORK_DIR}/next/ch_$i.dat"
fi
done
echo
echo
}
全個体番号の内、nextディレクトリにまだ居ない番号があれば、それをgenerate関数で乱数列で生成し直しています。
部分交叉は、今回筆者の考えで導入しました。実際には遺伝的アルゴリズムの世界にそのような交叉方法があるのかわかりません。
当初は「数列の各値で入れ替えが発生するか否かを1/2の確率で決める」という「一様交叉」で行っていました。しかし、世代を重ねても評価値が向上する様子がみられませんでした。
考えてみれば、機械語のバイナリ列は特定の1バイトを書き変えるだけでも動かなくなってしまうことさえあるので、現世代でせっかく良い評価であった個体も上位30%に入れず、交差する運命となった時点で全く別物に変わってしまうことになります。
各世代で交叉する際の変更は1バイトだけにして、少しの変更とその結果を評価することを繰り返すようにしようと考え、1バイトだけ入れ替える「部分交叉」を導入しました。
もっと考えてみると、実行バイナリと言えど、それは機械語という言語のCPUが解釈するソースコードと言えなくもないものです。普段のプログラミングで、(行き詰まったときなどは特に、)「少し書き換えて実行結果をみて、また少し書き変える」ということを繰り返していたりします。それを考えると、普段のプログラミングという行為を遺伝的アルゴリズムで表現するならば「部分交叉」という交叉方法になるのかな、と思います。
MBRテスターのtest_ch関数をすこし変更します。「"A"という文字を出力できたらpass」ではあまり面白くないので、「ASCIIの表示可能文字を出力できたらpass」というようにテストを変更し、遺伝的アルゴリズムではじめて出力する文字は何かを試してみます。
test_ch関数をリスト3.15のように変更します。
リスト3.15: ASCII表示可能文字を出力するかテスト
static uint8 test_ch(uint8 ch)
{
if ((0x21 <= ch) && (ch <= 0x7e)) {
printf("ch=%c(0x%02x)\n", ch, ch);
return 1;
} else
return 0;
}
変更後、コンパイルし、mbr_testerバイナリをge_mbr.shがあるディレクトリへ配置してください。また、mbr_testerはseabios.binとvgabios.binも必要とするため、同じディレクトリへ配置してください。
実行すると、図3.2のようにログが出力されていきます。
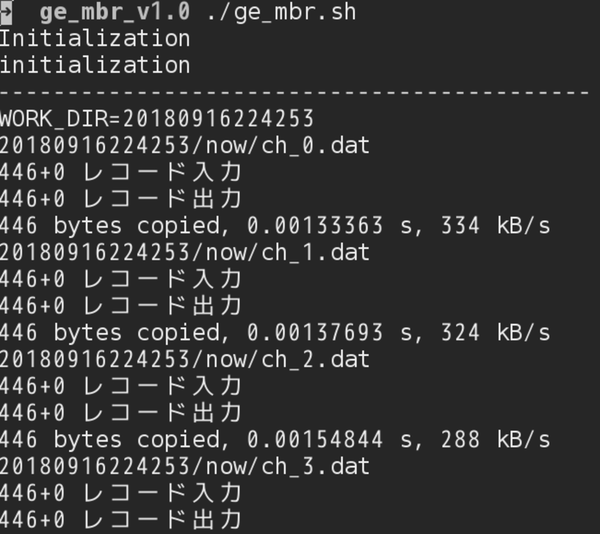
図3.2: ge_mbr.shの実行ログ
待っていると、、何やら文字を出力したようです!その文字は"B"みたいですね(図3.3)。
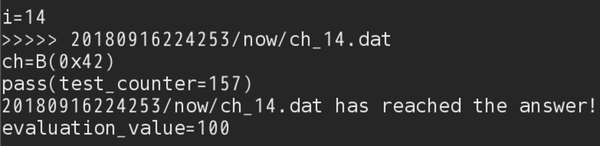
図3.3: 初めての文字は"B"
何を出力させていたのか、動作させて見てみます。前章にて、GUIに表示させる内容をコンソールに出力させるように改造したkvmulateで実行してみると、図3.4のようになっていました。
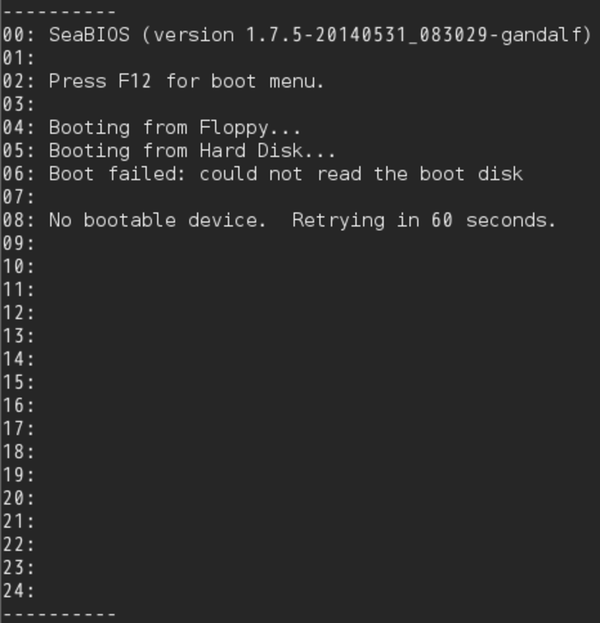
図3.4: "B"が出力されていた正体
MBRが出力していたのではなく、MBRはどうにかしてフロッピーディスクからのブートを失敗させて、BIOSにHDDからのブートを試させていたのでした。
「MBRに何らかの文字を出力するよう進化してほしい」という思惑からは外れましたが、「フロッピーディスクからブートする旨のメッセージが出力される4行目以降に何らかのメッセージを出力する」というテスト要件は満たしています。「ずるい手を使われたな」と思いつつも、神様の思ったとおりには動いてくれないものだな、となんだか面白い結果でした。
やってみると分かりますが、テストを適切に用意してあげないと、今回の様にちょっとずるい手に走ってしまったりします(それはそれで面白いのですが)。
そもそも0点か100点しかない評価方法だと、100点の個体が生まれるまで全員0点ということになり、「優れたものを残し、掛け合わせることで答えに近づける」という流れにできません。やはり評価としてはpass/failではなく、点数を返すような評価方法じゃないといつまでたっても成長しないですね。(当たり前のことかもしれませんが。。)
そこで、数値的に評価できる指標として、「画面を描画したピクセル数」を評価値としてみます。
MBRテスターとge_mbr.shを変更します。変更箇所の要点だけ説明すると、以下の通りです。
詳細は、MBRテスターの差分については筆者のウェブページを*3、ge_mbr.shはリポジトリのtest_fillブランチ*4を見てみてください。
[*3] http://yuma.ohgami.jp
[*4] https://github.com/cupnes/ge_mbr/tree/test_fill
紹介したいのはこの結果です。
ge_mbr.sh実行後、最初のころは、BIOSが出力するメッセージ分のピクセル数(5,000pixel程度)がほとんどの個体の評価値でした。
628世代で評価値が急に13,000pixelへ上がりました。1つ目の進化です。これは、BIOSが大量のエラーログを吐き出しているためで、パターンとしては前述の「'B'を出力した」時と同じです。そしてこれを超える進化は一向に起こらず、この個体の繁栄が続きます。
驚くべきは2510世代で、この世代の19番目の個体(ch_19.dat)の評価をする際、MBRテスターが終了せずに固まっていました。画面をよく見ると、文字ではなくカラフルなドットが描画されていました。何が起きたのかというと、ASCIIをVRAMへ書き込んで文字を出力する「テキストモード」から、VRAMへピクセルフォーマットに従ったピクセルデータを書き込んで描画する「グラフィックモード」へビデオモードを変更していたのでした。「一定時間待って描画ピクセル数を数え、終了する」処理は「updateTextmode関数」側に入れていたため、グラフィックモードに移行するとVMであるMBRテスターが終了せず、そのまま固まっていたのです。
これは偉大な進化です。しかし、この時は固まったMBRテスターをkillで終了させるしかなく、その場合、この個体の評価値は0となるため、次の世代で淘汰されたのか、居なくなってしまいました。ただ、過去の個体データはすべて残っているため、過去の個体データから実行を再開し、同じ状況を再現したのが表紙の写真です*5。
[*5] なお、表紙は「2001年宇宙の旅」が2001年にアメリカで再上映された際のポスターが元ネタです。元ネタのポスターでは、MBRを実行しているウィンドウのあたりにスターチャイルドが居て、こっちを見ています。それにならって、進化したMBRがこっちを見ているイメージです。