第2章 設計とモデル
前章で説明した「5つの振る舞い」と「細胞の構造」を設計しUMLでモデル化します。
そして、本書でPoC*1実装するバイナリ生物とその処理系がその中でどの様に実現されるのかを説明します。
[*1] 「Proof of Concept(概念実証)」の略。本書でサンプルとして紹介する実装は「生きている」と言い張る事を満たしてはいますが、それ以上のものではありません。
2.1 PoC実装するバイナリ生物について
本書では、「インクリメンタ細胞」という細胞一つを体の本体とする「インクリメンタ生物」という単細胞生物を作ります。
インクリメンタ生物は以下のようにして生きている生物であるとします。(「細胞」や「化合物」等をどのようにモデル化するかは後述しますので、ここでは「そのようなものか」と思っておいてください。)
- 化合物(64ビット整数値)を取り込み、細胞内のタンパク質と結合して代謝を行うことで生きている
- 代謝により化合物はインクリメントされた値へ変質する
- インクリメント後の化合物は体外へ排出する
それでは、次節から、前章で説明した構造と振る舞いの設計とモデル化を説明します。
2.2 細胞の構造を掘り下げる
前章で紹介した「タンパク質」・「DNA」・「属性情報(寿命)」について、バイナリの世界にどのように対応付けるのかを掘り下げて説明します。
そして、この節の最後に、細胞の各構成要素の機能とそれぞれの関係をクラス図で示します。
タンパク質: バイナリの世界との対応付け
前章で、細胞とその構成要素であるタンパク質の構成を以下のように説明しました。
- 「細胞」は、複数の「タンパク質」で機能を果たす
- 「タンパク質」は、複数の「化合物」でできている
- 「化合物」は、複数の「元素」でできている
- 「タンパク質」は、複数の「化合物」でできている
これを、バイナリの世界における「関数」の以下の構成と対応付けて考えます。
- 「関数」は、複数の「機械語命令」で機能を果たす
- 「命令」は、1つあるいは複数の「命令の構成要素」でできている
- 「命令の構成要素」は、1つあるいは複数の「1バイトデータ」でできている
- 「命令」は、1つあるいは複数の「命令の構成要素」でできている
「命令の構成要素」とは、主に「オペコード」・「オペランド」等と呼ばれる1つの命令列を構成する要素です。タンパク質と命令を図2.1の様に置き換えて考えてみるわけです。
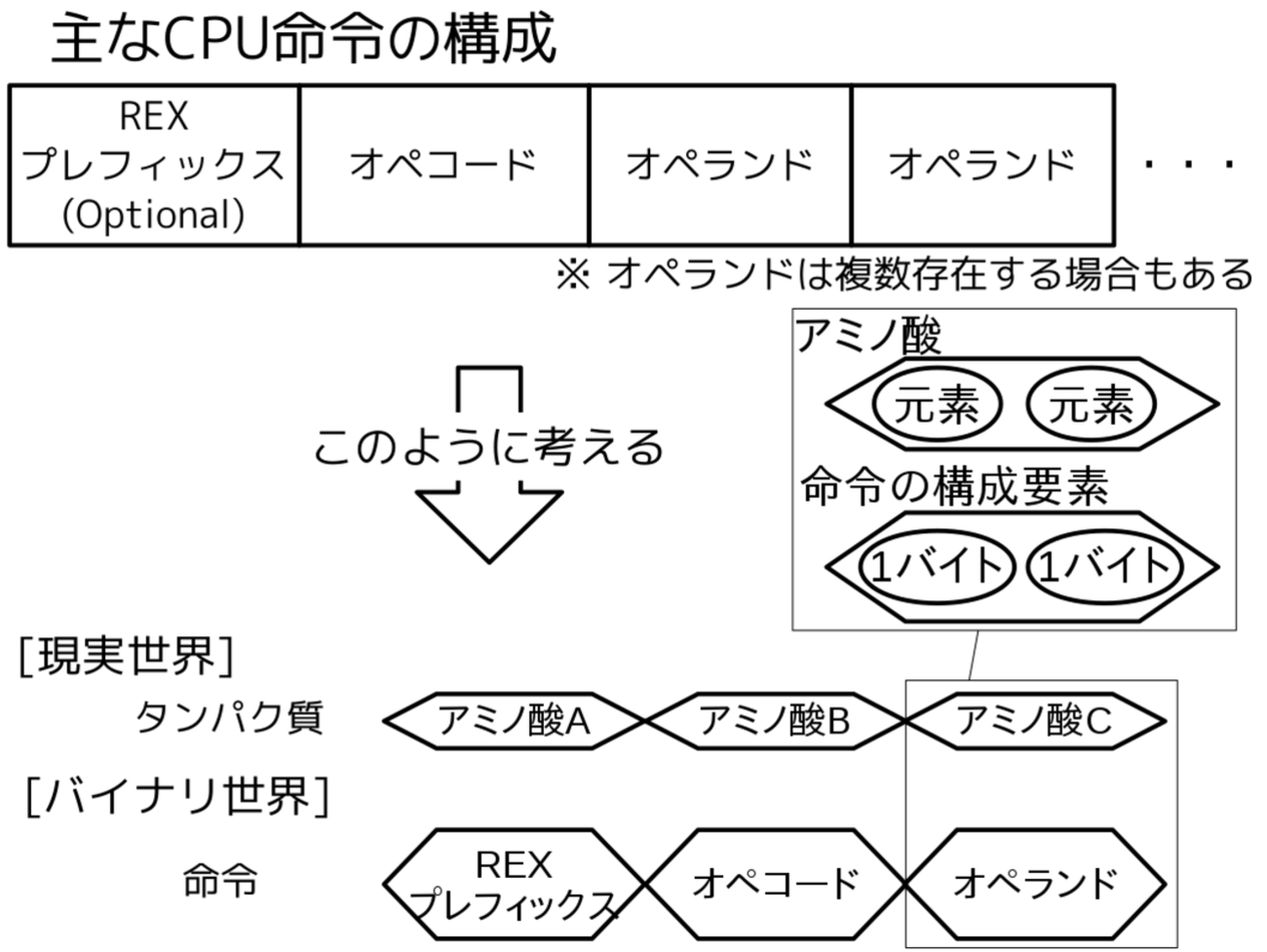
図2.1: CPU命令とタンパク質の関係
以上を踏まえて、現実世界とバイナリ世界の対応をまとめると図2.2の通りです。
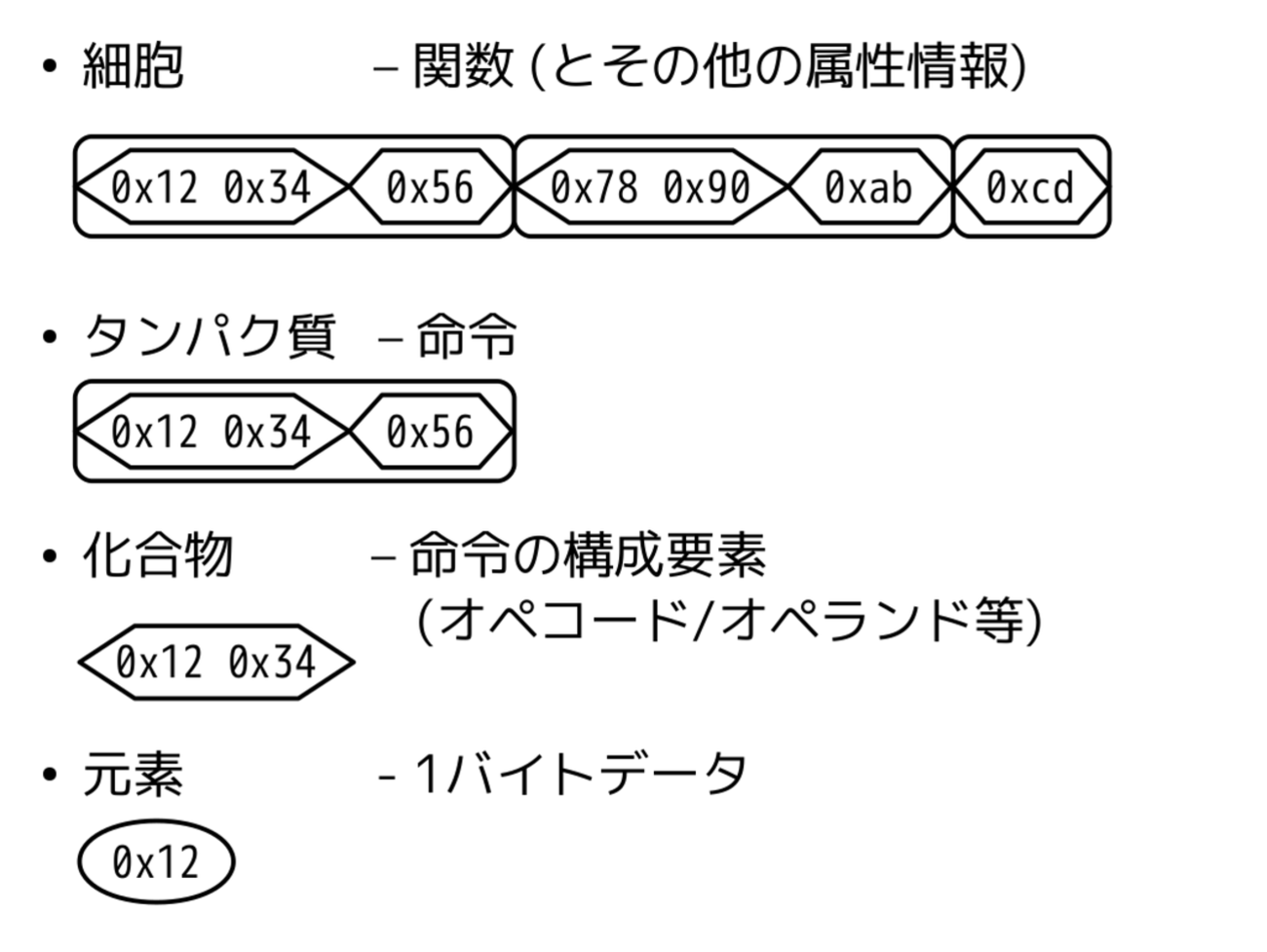
図2.2: 現実世界とバイナリ世界の対応まとめ
すなわち、バイナリ世界における「単細胞生物」は、「関数1つだけの実行バイナリ」となります。
そして、細胞の「タンパク質が触媒となり物質を別の物質へ変質させる(代謝)」に対して、バイナリ世界でも「関数実行により引数が戻り値へ変質している」と考え、「関数実行」を「代謝」と対応付けます。また、細胞は「代謝の際に得られたエネルギーを使って何らかの動作を行う(運動)」のですが、これも「関数実行により何らかの処理が実施される」と対応付けることで、「運動」も「関数実行」に含まれると考えます。
また、ここで、「化合物」には「命令の構成要素になるもの」と「関数の引数になるもの」の2種類が存在することになります。本書では、命令の構成要素になる化合物を「コード化合物」、関数の引数になる化合物を「データ化合物」と呼ぶことにします。
タンパク質: インクリメンタ細胞を表現する
以上を踏まえ、インクリメンタ細胞ではどの様に表現するのかを考えます。
まず、インクリメンタ細胞は、「引数(64ビット整数値)をインクリメントして返す関数(インクリメント関数)」だけで構成された実行バイナリとします。
インクリメント関数はC言語で書くとリスト2.1のように書けます。
unsigned long long incrementer(unsigned long long num)
{
return num + 1;
}
これをGCC等でコンパイルすると7命令程の機械語バイナリになるのですが、「インクリメントして返す」だけであれば3命令で実現できます(リスト2.2)。
mov %rdi, %rax inc %rax ret
簡単に説明すると、C言語の関数の第1引数は、コンパイル後の機械語バイナリ(およびアセンブラ)では、RDIというレジスタで扱われます。そのため、RDIレジスタの内容を確認すれば、呼び出し元が第1引数で渡してきた値を確認できます。
ここでは、最初の「mov」命令でRDIレジスタの中身をRAXというレジスタへコピーします。
次に、「inc」命令でRAXレジスタの中身をインクリメントします。
最後に、「ret」命令で呼び出し元へジャンプします。その際、RAXレジスタが戻り値となるので、引数で渡した値がインクリメントされて返されることになります。
これはアセンブラでの表現なので、これをCPUが直接解釈する機械語に置き換える(アセンブル)とリスト2.3の通りです。これが機械語バイナリ(の実行コード部分)となります。
0x48 0x89 0xf8 0x48 0xff 0xc0 0xc3
リスト2.3の各命令を、命令の構成要素で分解し、細胞の構造と対応付けるとリスト2.4の通りです。
mov命令(タンパク質): - REXプレフィックス(化合物): 0x48 - オペコード(化合物) : 0x89 - オペランド(化合物) : 0xf8 inc命令(タンパク質): - REXプレフィックス(化合物): 0x48 - オペコード(化合物) : 0xff - オペランド(化合物) : 0xc0 ret命令(タンパク質): - オペコード(化合物) : 0xc3
「REXプレフィックス」は「64ビット命令」を示す1バイトです。例えば、「0x48 0x89 0xf8」を「0x89 0xf8」と書いた場合、「mov %edi,%eax」という32ビットの命令になります*2。
[*2] 詳しい説明は省きますが、先頭に"R"が付く「RDI」等のレジスタは64ビットのレジスタで、先頭に"E"が付く「EDI」等のレジスタは32ビットのレジスタです。この例では、REXプレフィックスを付けることでmov対象のレジスタのビット幅が変わっています。
以上から、本書でPoC実装する世界には6種のコード化合物と、特に種別の無い8バイトのデータ化合物が存在することになります。
DNA: バイナリの世界でどのように表現するか
本書では、DNAはコドンが連結したリストとして扱います。本書で扱うバイナリ生物の世界観ではDNAの構造の最小単位をコドンとし、ヌクレオチドに相当する粒度は扱いません*3。
[*3] 本書では実装の簡単さ等も考えてこのようにしましたが、DNAのモデル化はもう少し良い方法があるのかもしれません。
タンパク質の説明の際は、現実世界と対比させて説明しましたが、プログラムのバイナリ世界のDNAは現実世界と結構変えてしまったので、モデル化した結果のみ説明します。バイナリ世界のDNAを図示すると図2.3の通りです。
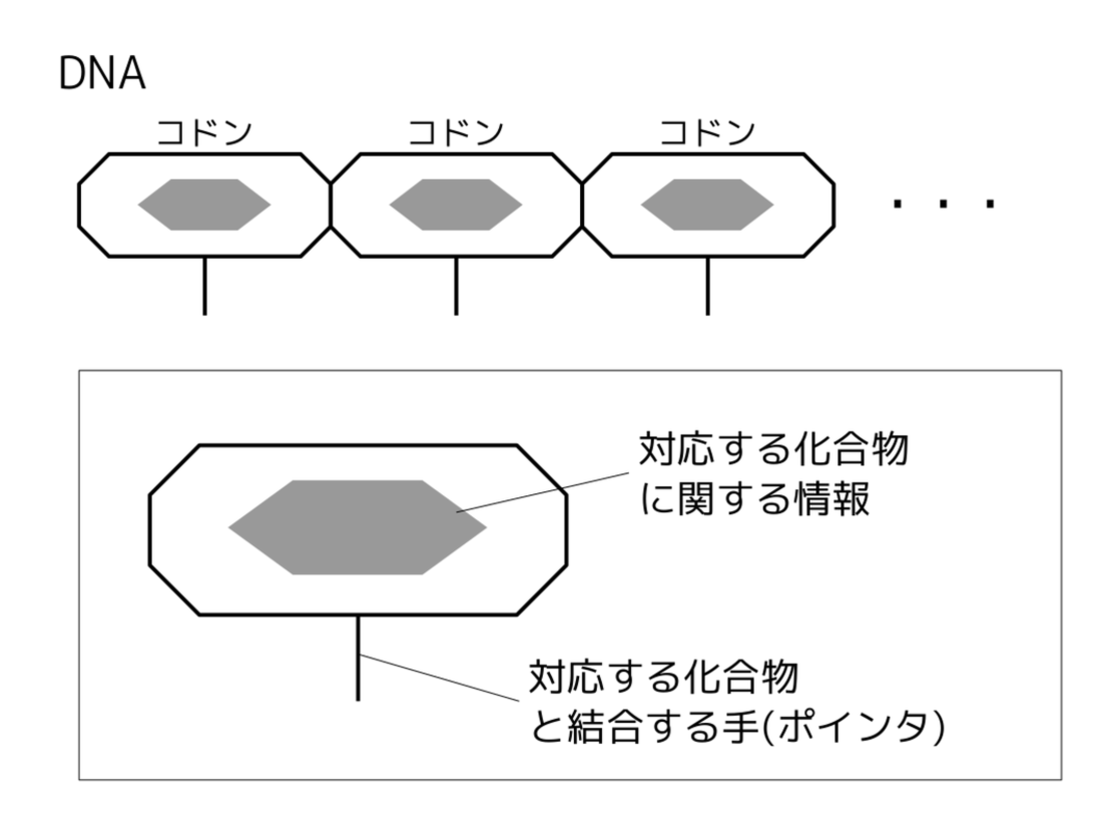
図2.3: バイナリ世界のDNA
各コドンは対応するコード化合物に関する情報を持ちます。コード化合物、すなわち命令を構成するオペコード/オペランド等の要素は、それぞれの長くとも64ビットを越えることは無いので、コドンは対応するコード化合物を64ビット整数値で保持することができます。
そして、コドンに対応するコード化合物を取り込んだときに、それと結合しておくための手として化合物へのポインタも持ちます。
このポインタを使用して図2.4のようにセントラルドグマを行い、細胞を構成するタンパク質を生成します。
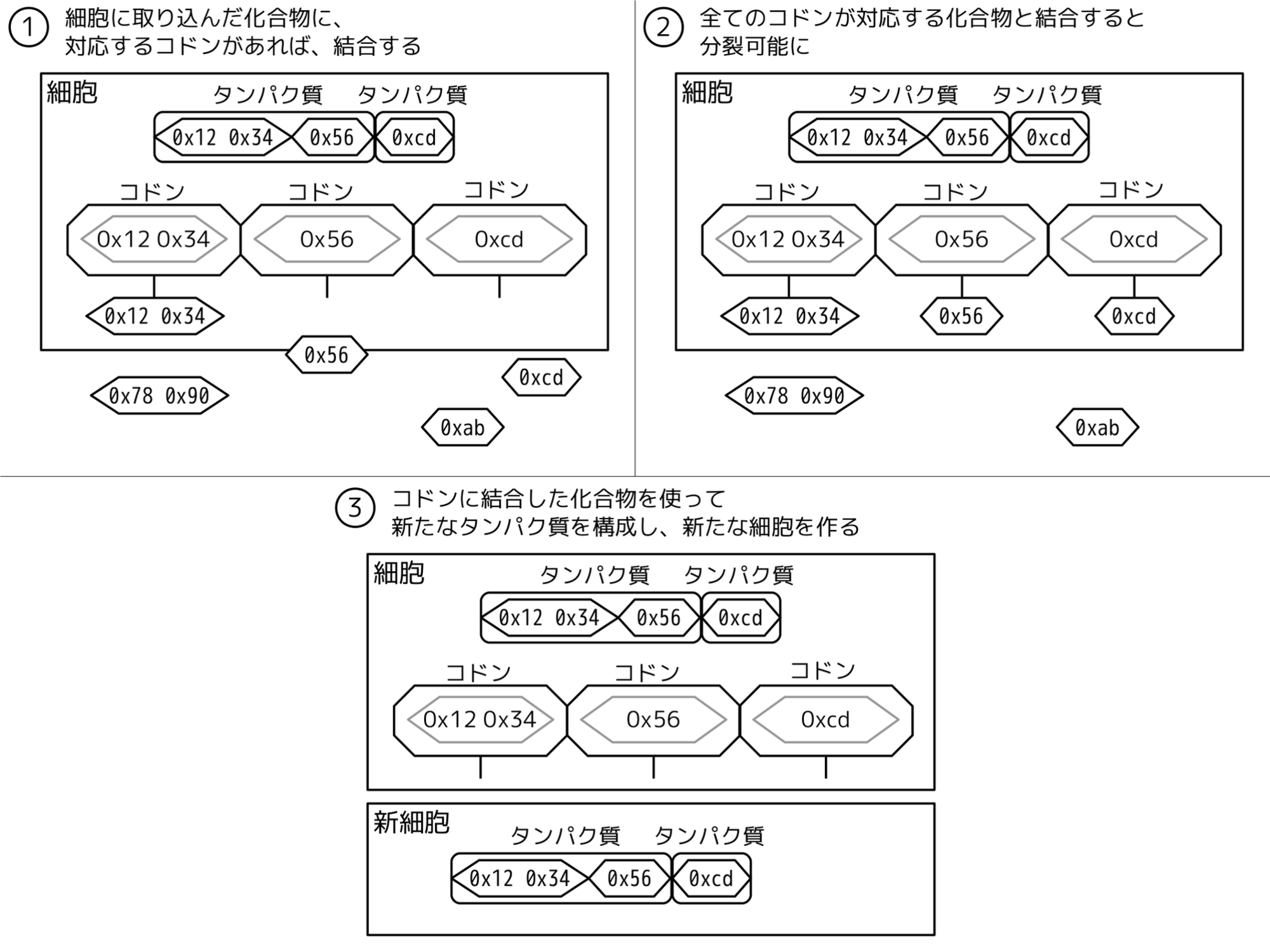
図2.4: バイナリ世界のセントラルドグマ
属性情報(寿命): バイナリの世界でどのように表現するか
属性情報として寿命は、細胞のデータ構造の中に整数値として持たせておき、周期毎にデクリメントすることにします。
そして、0になると、「死」の振る舞いを実施することにします。(振る舞いの内容は後述します。)
バイナリ生物の細胞の構造まとめ
これまで説明してきたことを、それぞれの構造の関係を示すためにUMLのクラス図で図示してみると図2.5の様になります。なお、各構造が持つ関数は、各構造の役割として必須なものを挙げているだけで、これが全てというわけではありません。(特にそれぞれの構造で内部的に使う関数などは、実装の仕方により増えることもあると思います。)
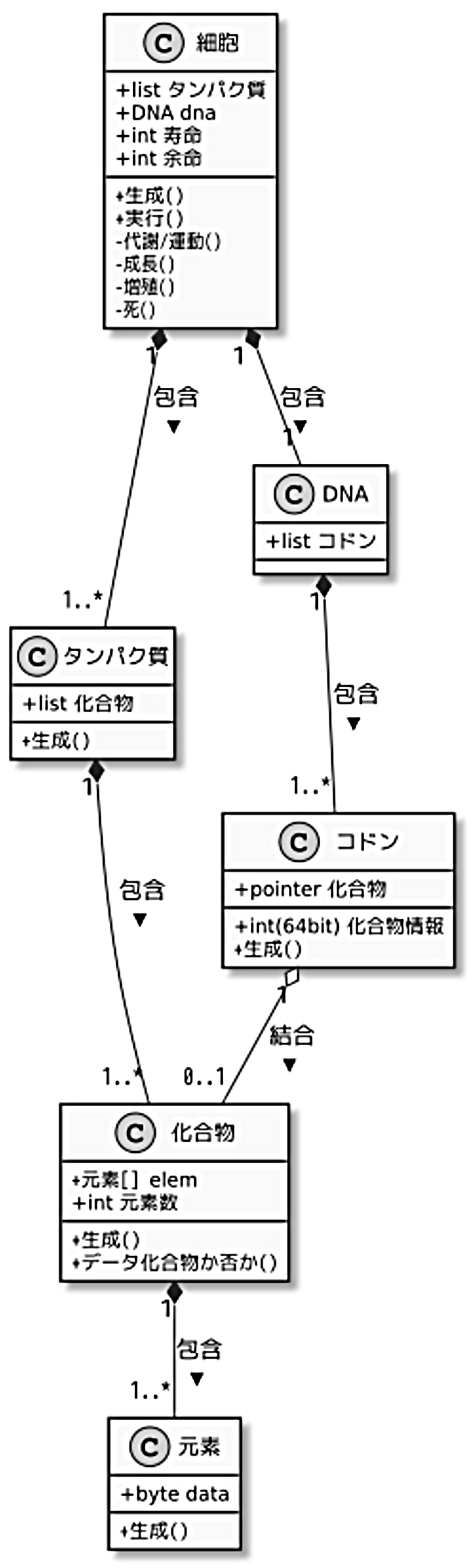
図2.5: 細胞から元素までの構造
以上、大分ざっくりとした説明ですが、複雑にモデル化すればするほど、それを実装する必要があるので、ひとまず簡単な単細胞生物をバイナリ生命体として実現する上では以上のモデルで十分です。
2.3 処理系
ここまで、実行バイナリを生物として対応付けて考える事を説明してきました。ただし、そのためには、実行バイナリを先述の構造に当てはめる形でロードし、後述する生物としての5つの振る舞いを行わせる「処理系」が必要です。
そのために、「環境に存在する細胞と化合物を管理する」、「細胞の要求に応じて環境に存在する化合物を与える/あるいは環境へ配置する」、「周期を管理し各周期で環境に存在する全ての細胞の1周期を実施する」などが必要です。
なお、実行バイナリをロードする部分は本書のPoCでは扱いません。PoC実装では、処理系の初期化時に実行バイナリをロード済みの状態で細胞やタンパク質の初期状態をセットアップします。
振る舞いのモデルは次節で紹介します。
2.4 処理系と5つの振る舞いを考える
前節までで設計した構造を元に、バイナリ生物における細胞とその処理系の振る舞いを考えます。
処理系
前述した構造で用意された細胞を処理する処理系としては図2.6のような振る舞いをするものを考えます*4。
[*4] なお、PoC実装の対応するソースコードの箇所はリポジトリ直下main.cのmain関数です。適宜参照してみてください。
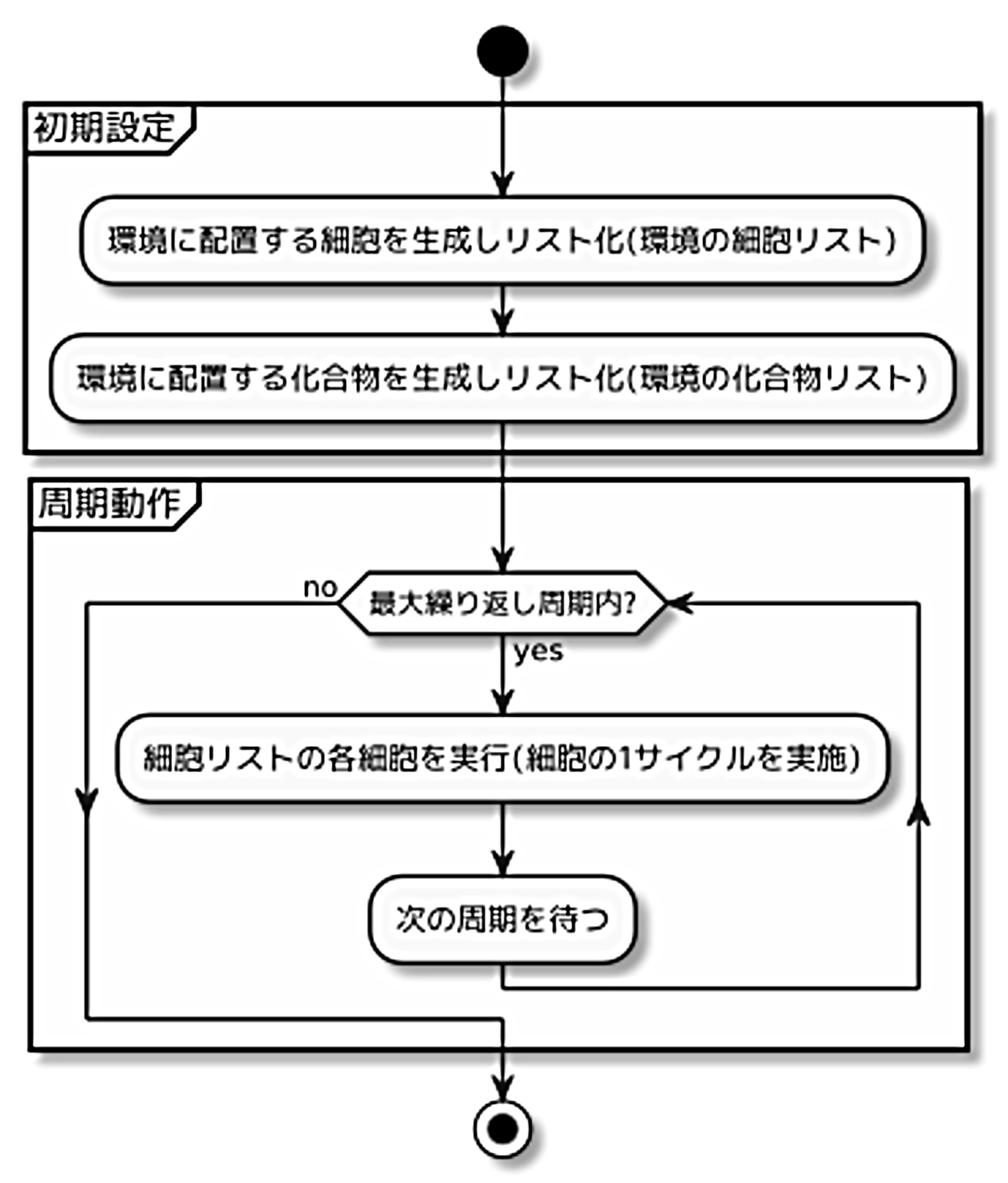
図2.6: 処理系の振る舞い
前述した通り、今回のPoCサンプルでは実行バイナリをロードする部分は扱いません。処理系の初期設定時に細胞の構造を構築します。
初期設定を終えた後は、細胞のリストに存在する細胞を毎周期で全て実行します。
細胞の1周期
次に、細胞の1周期です。1周期の中で、「代謝/運動」・「成長」・「増殖」・「死」の振る舞いが適宜呼び出されます(図2.7)*5。
[*5] PoC実装の対応箇所はリポジトリ直下cell.cのcell_run関数です。
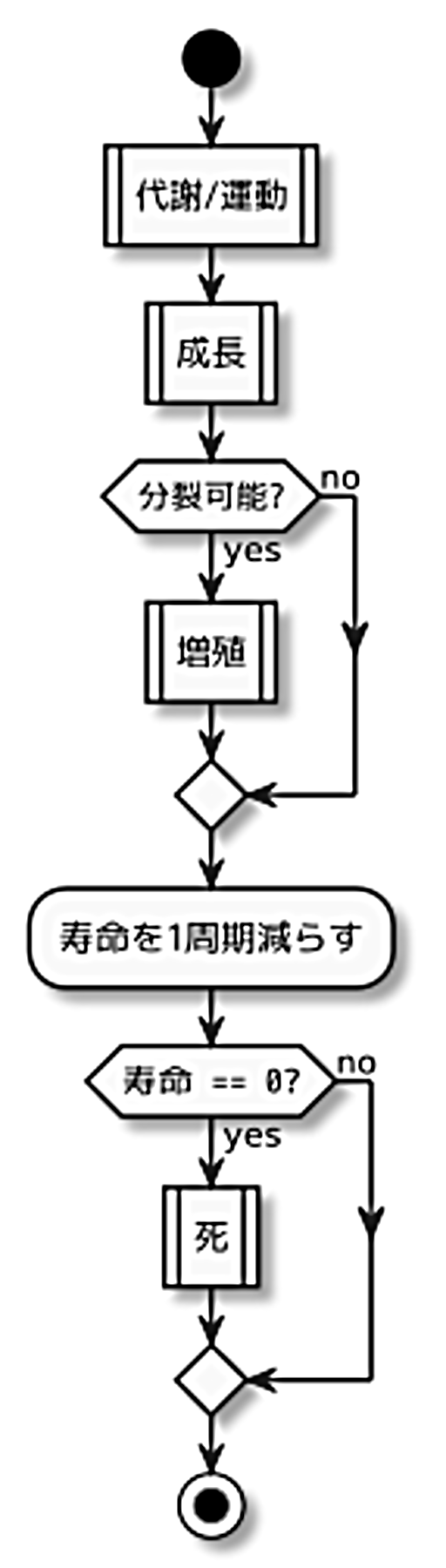
図2.7: 細胞の1周期の振る舞い
毎周期で「代謝/運動」と「成長」を実施し、成長後、分裂可能であれば「増殖」を行います。寿命も毎周期で減っていき、0になると「死」の処理を実施します。
以降で、「代謝/運動」・「成長」・「増殖」・「死」それぞれの振る舞いを紹介します。
代謝/運動
「代謝/運動」の振る舞いを図示すると図2.8の通りです*6。
[*6] また、PoC実装の対応箇所はリポジトリ直下cell.cのmetabolism_and_motion関数です。
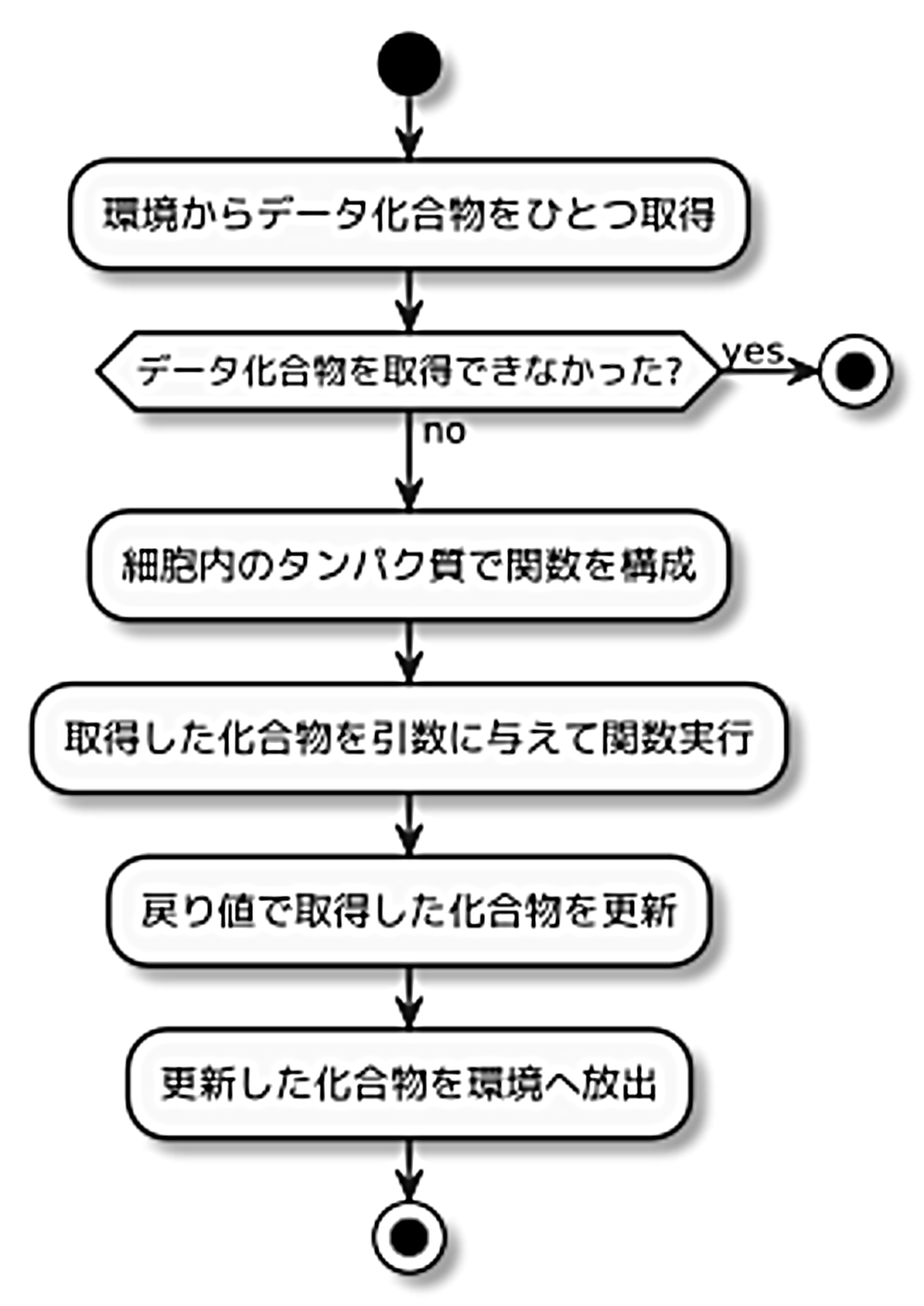
図2.8: 代謝/運動の振る舞い
まず、環境から関数の引数となるデータ化合物をひとつ取得します。もし取得できなければこの周期での代謝/運動は何もしません。
データ化合物を取得できた場合、細胞内の各タンパク質を辿って化合物を取得し、メモリ上で各化合物の内容を結合して関数を作ります。そして、取得したデータ化合物の内容を引数として関数を実行します。前述した通り、この関数実行が代謝と運動に当たります。
最後に、関数の戻り値でデータ化合物を更新し、環境へ戻します。
成長
関数を構成するオペコード/オペランド等のバイト列(化合物)を集める作業を「成長」と考えます。「成長」の振る舞いを図示すると図2.9の通りです*7。
[*7] PoC実装の対応箇所はリポジトリ直下cell.cのgrowth関数です。
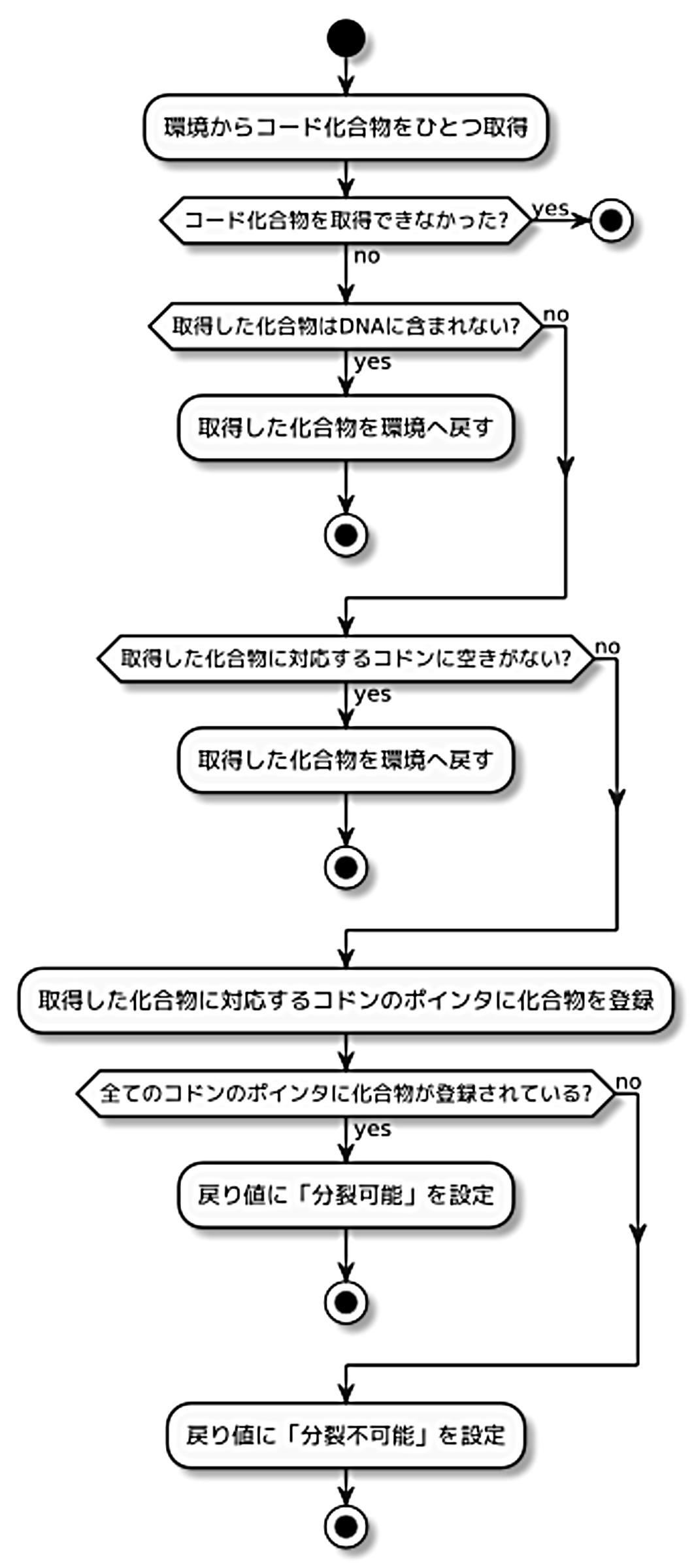
図2.9: 成長の振る舞い
まず、環境からコード化合物を取得します。取得できなかったり、取得したがDNAに含まれていなかったり、DNAに含まれているが既に取得済みである場合は、適宜取得した化合物を環境へ戻した後、何もせずこの周期の成長を終えます。
取得した化合物がDNAに含まれ、それをまだ取得していなかった場合、DNA内の対応するコドンが持つポインタに取得したコード化合物を設定します。
その後、DNA内の全てのコドンに化合物が設定済みか否かをチェックし、設定済みである場合は分裂可能を示す値を、未設定のコドンが一つでもあれば分裂不可能を示す値を返します。
増殖
細胞における増殖は細胞分裂です。DNAのコドンに結合された化合物を使って、新たに確保した構造へ自身のコピーを作ることに当たります。
[*8] 対応するPoC実装はリポジトリ直下cell.cのdivision関数です。
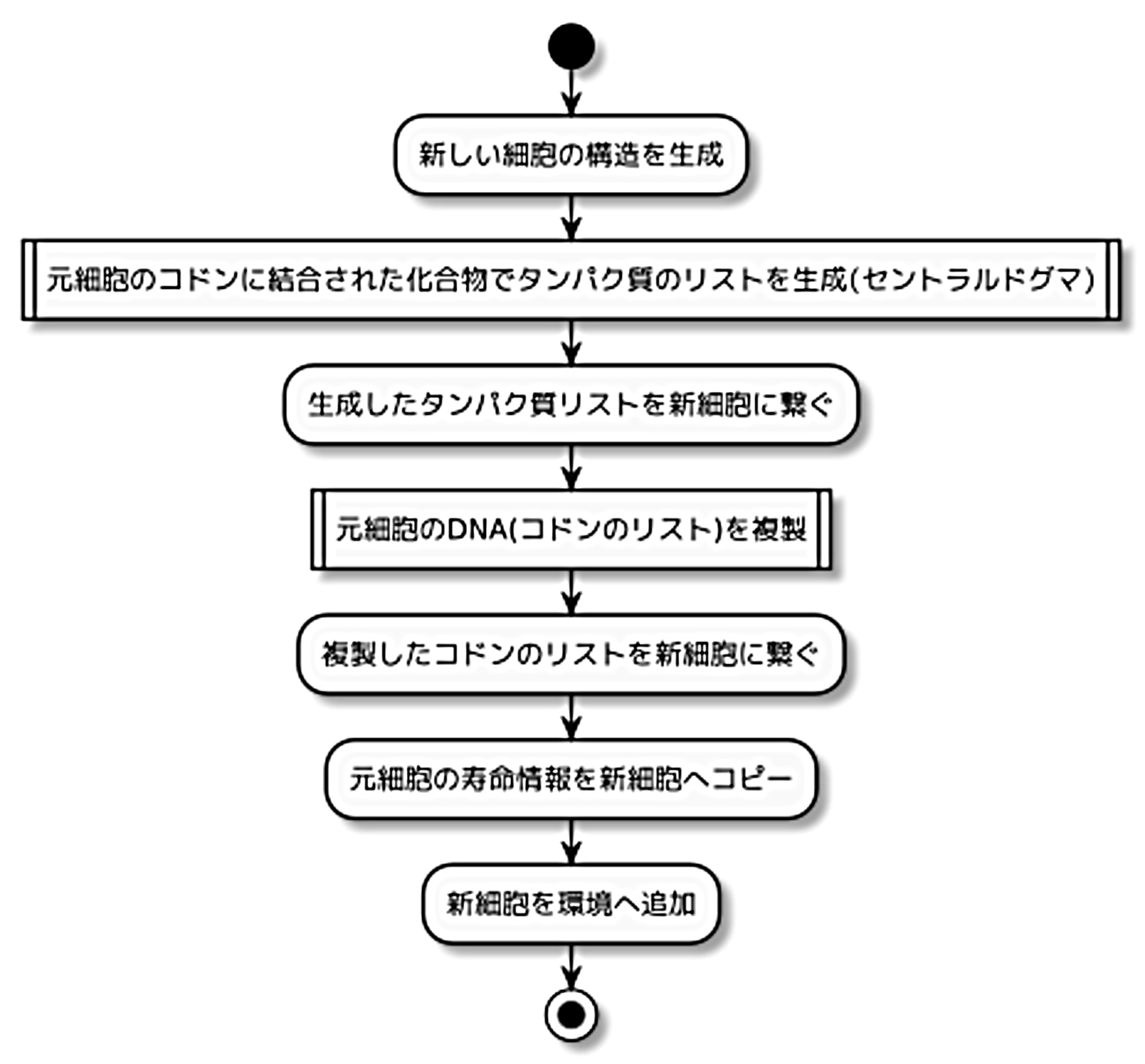
図2.10: 増殖の振る舞い
新しい細胞を中身を空で生成した後、セントラルドグマにより元細胞のDNAからタンパク質リストを生成し、新細胞のタンパク質リストへ繋ぎます。
その後、コドンも中身を空で生成し元細胞のDNAのコドンをそこへコピーした後、コドンのリスト(DNA)を作成し、これも新細胞へ繋ぎます。
最後に元細胞の寿命情報を新細胞へコピーして、できあがった細胞を環境へ追加すれば終わりです。環境へ追加すれば、以降は処理系により実行されるようになります。
死
細胞においてもバイナリ世界においても、死はリソースの解放です。自身を構成している部品を環境へ解放します。
[*9] 対応するPoC実装はリポジトリ直下cell.cのdeath関数です。
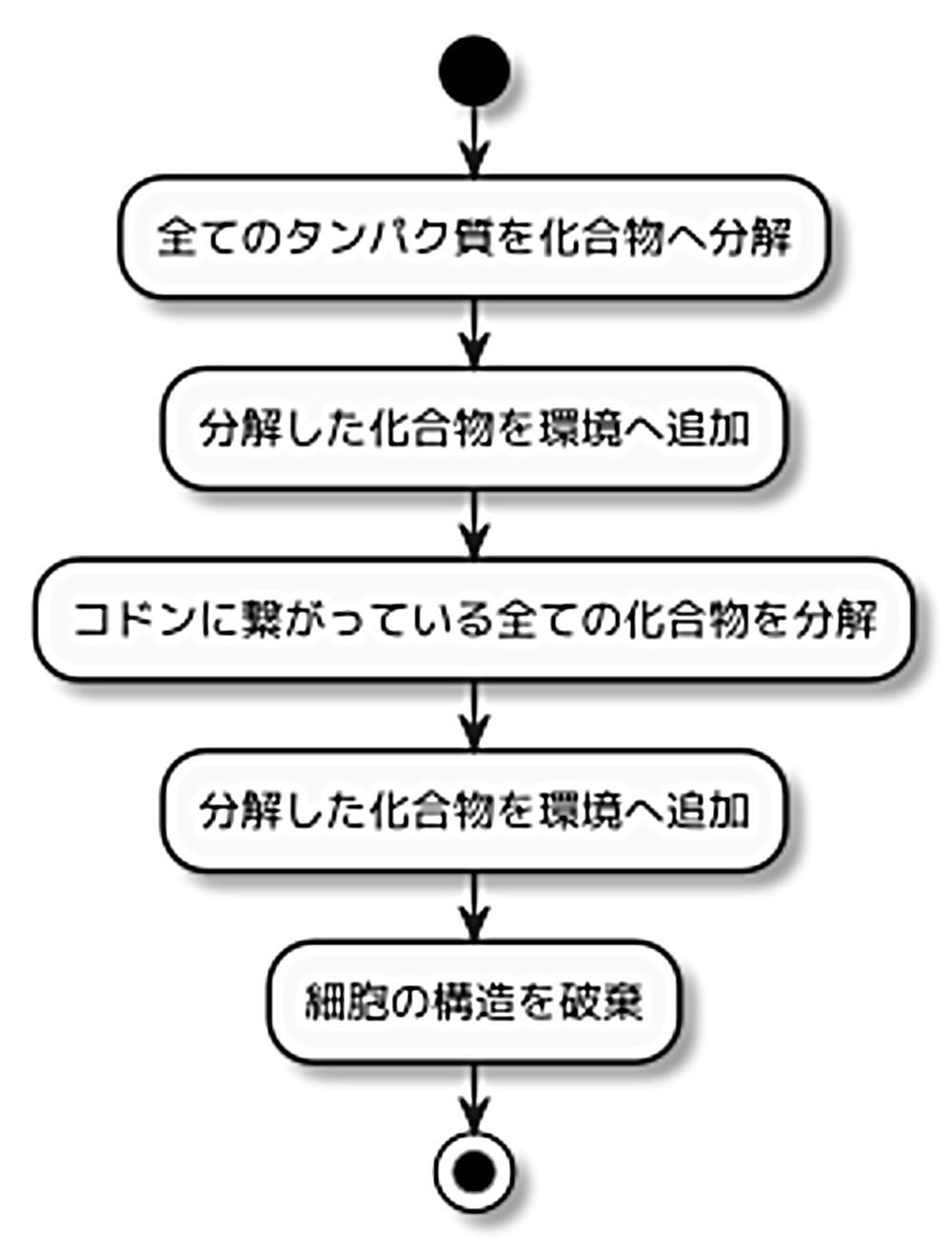
図2.11: 死の振る舞い
死の際に細胞が分解して環境へ放出する要素は以下の2つです。
- タンパク質: 化合物のレベルまで分解し、環境へ放出
- DNA: コドンに結合している化合物があれば全て分離し、環境へ放出
これら2つの要素を環境へ放出した後は、細胞の構造を破棄します。
2.5 まとめ
この章では、バイナリの世界で細胞をどのように表現するか、その処理系も含め、設計とモデルを説明しました。ポイントをまとめると以下の通りです。
- 「細胞」はプログラムにおける「関数」に対応すると考える
- 「タンパク質」は「命令」に対応し、「化合物」は「命令の構成要素」、「元素」は「バイト」に対応する
- この構造を用いて、生物の「5つの振る舞い」を置き換えてモデル化した
また、本書のPoC実装では、化合物をインクリメントして返す「インクリメンタ細胞」をもつ生物を作成します。この章で、この細胞の場合にどうなるかを適宜説明しました。