第3章 PoC実装の実行結果
3.1 PoC実装について
「代謝/運動」・「成長」・「増殖」・「死」の振る舞いを実際に確認するPoC実装を行ってみました。
このPoC実装では、前章のモデルを処理する処理系を実装した上で、その処理系の環境内に前章で紹介した単細胞生物「インクリメンタ」を1つ配置し、以下の振る舞いを確認します。
- 細胞が持つタンパク質(命令)リストからメモリ上に関数を動的に構成し、同じく動的に取得した引数に対して処理を実行できる(代謝/運動)
- 環境内に配置したコード化合物の内、DNAに合致するものを動的に取り込む(成長)
- DNAに記載されたコード化合物を全て揃えると細胞分裂が行われる(増殖)
- 寿命を迎えるとタンパク質内の化合物とDNAのコドンが持つ化合物への参照を分解し環境へ放出する(死)
実装の特筆事項
実装の内容については本書で解説は行いません。設計は前章でモデル図も用いて紹介したとおりで、それをどのように実装するかは好み次第なところもあるので、もし興味があれば本書のサンプルリポジトリをご覧ください。
ここでは、実装に関する特筆事項のみ説明します。
まず、実装は筆者の趣味もあり、筆者の自作OS上のアプリケーションとして実装しています。本書で引用するスクリーンショットも自作OS上で動作しているものです。
ただし、前章でモデル図などで説明した設計は、Linux等でも実装可能な内容ですので、もし(万が一、物好きにも。。)興味があれば、ぜひご自身で実装してみてください。
今回のPoC実装では、主に簡単化のために以下の方針で実装しました。
- 実行バイナリはファイルからロードするのではなく、処理系の中で初期化時にメモリ上に作る
- 「DNA」という独立した構造は用意していない
- 細胞がコドンのリストを持つ形にした
- DNAという中間層は現状では実質機能しないためです
- 細胞/タンパク質/化合物/コドンのそれぞれは、予め中身が空の構造をグローバル変数でいくつか用意しておき、それを使い回します
- 簡単のため(手抜きのため)です。。
3.2 PoCプログラムの実行結果と評価
PoCプログラムを実行すると図3.1の画面が表示され、自動で進んでいきます。
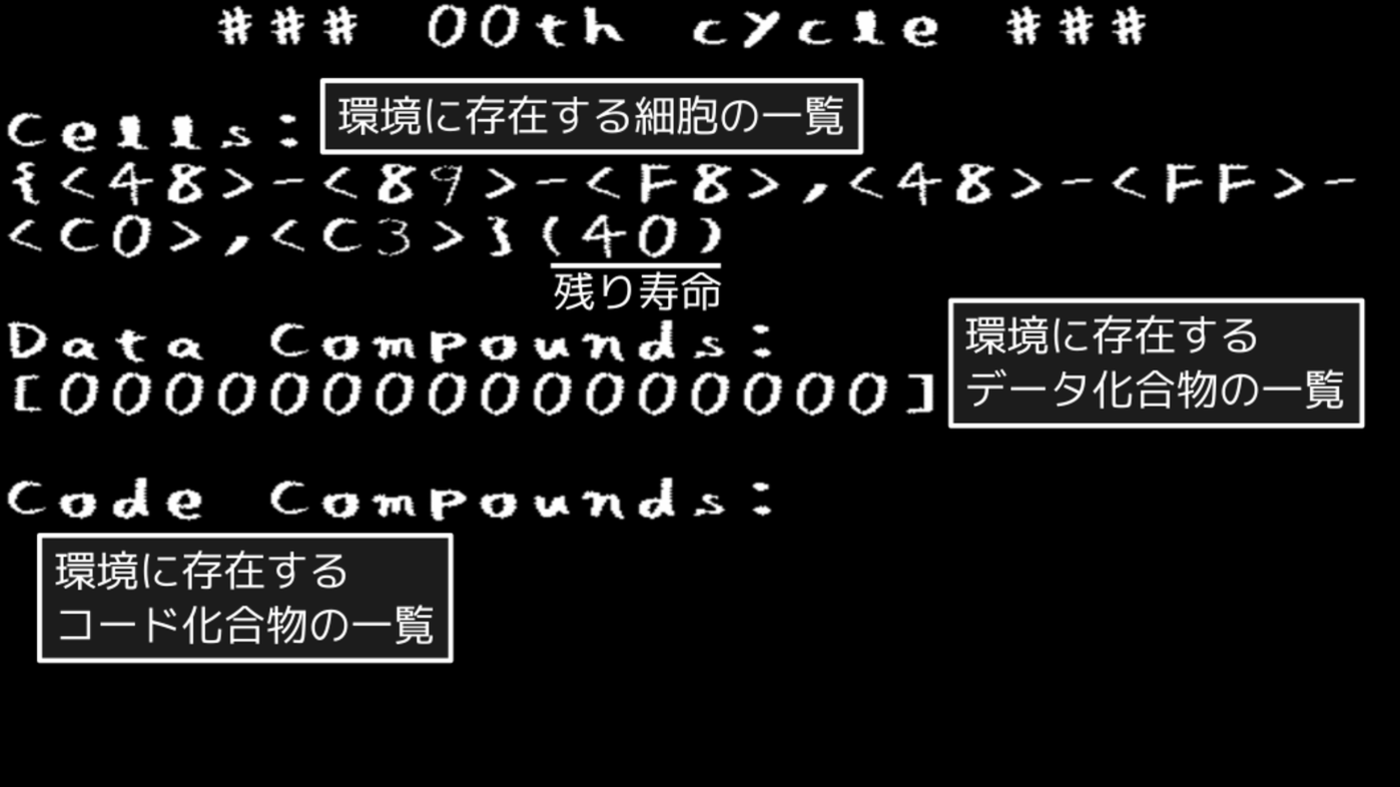
図3.1: PoCプログラム起動直後の画面
画面には現在の周期(「XXth cycle」の箇所)と、環境に存在する細胞(「Cells:」の箇所)と、データ化合物・コード化合物(「Data Compounds:」と「Code Compounds:」の箇所)が表示されていて、1秒周期で進んでいきます*1。この画面で、以下の要素が現在の環境に存在することを示しています。
[*1] フレームバッファの制御を作り込んでいないため、特に実機で試す際はチラツキますがPoC程度ということで気にしないでください。あるいは画面の更新箇所を減らすなど工夫してみてください。
- 細胞
- インクリメント関数の細胞(単細胞生物)
- 寿命は40周期
- 化合物
- データ化合物
- 8バイト整数
- 初期値0
- コード化合物
- 無し
- データ化合物
周期が進む毎にインクリメント細胞によりデータ化合物がインクリメントされていき(代謝/運動)、細胞自身の寿命も減っていきます(図3.2)。
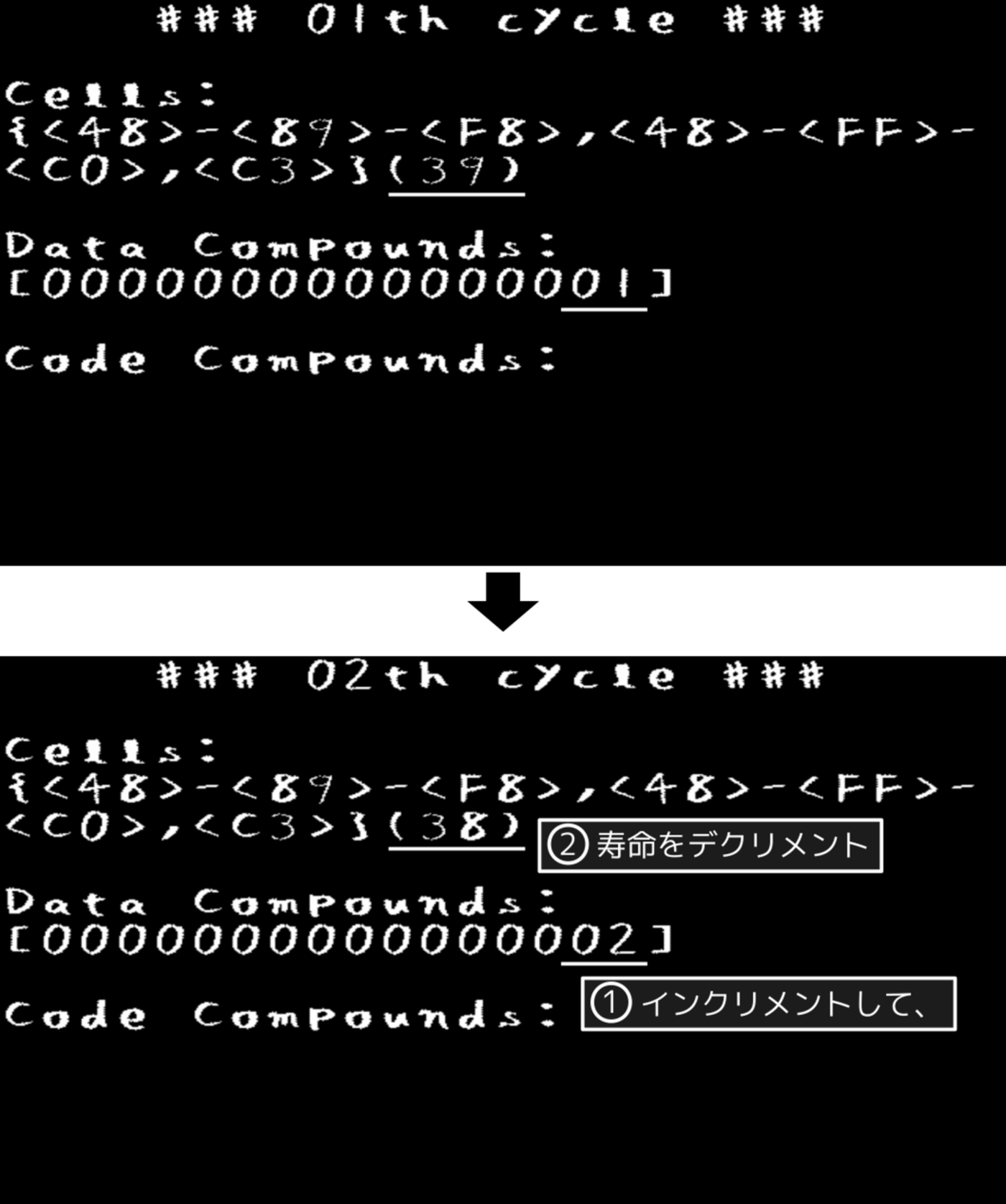
図3.2: PoCプログラムの実行画面(周期01 - 02)
10周期目に、インクリメント細胞のタンパク質を構成する(すなわち、細胞のDNAに記録されている)全てのコード化合物が環境に投与されます。すると、インクリメント細胞は1周期に一つずつ化合物を取り込んでいきます(図3.3)(成長)。
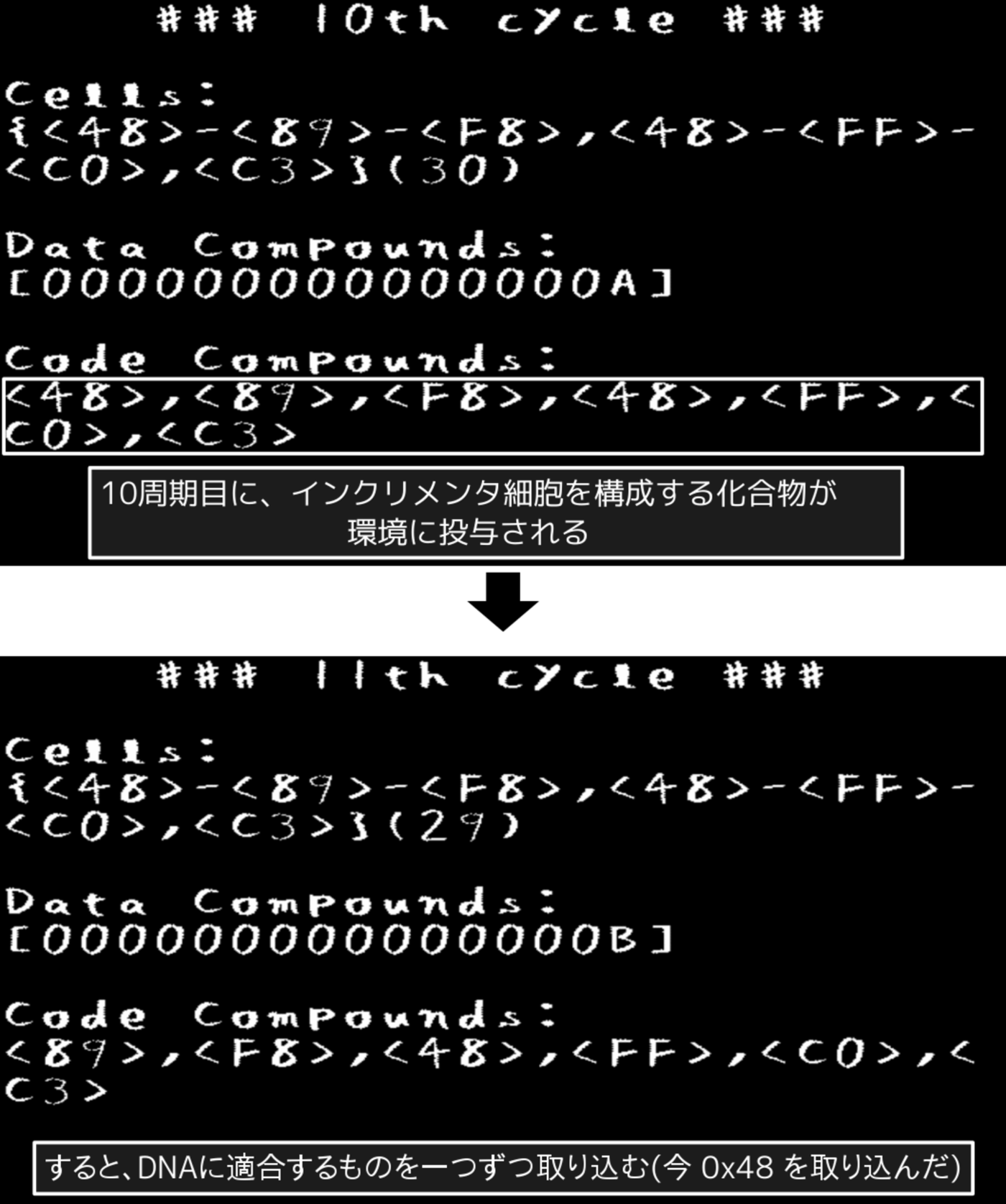
図3.3: PoCプログラムの実行画面(周期10 - 11)
全ての化合物を取り込むと、DNAに記載された化合物が全て揃うので「分裂可能」となり、細胞分裂を行います(図3.4)(増殖)。
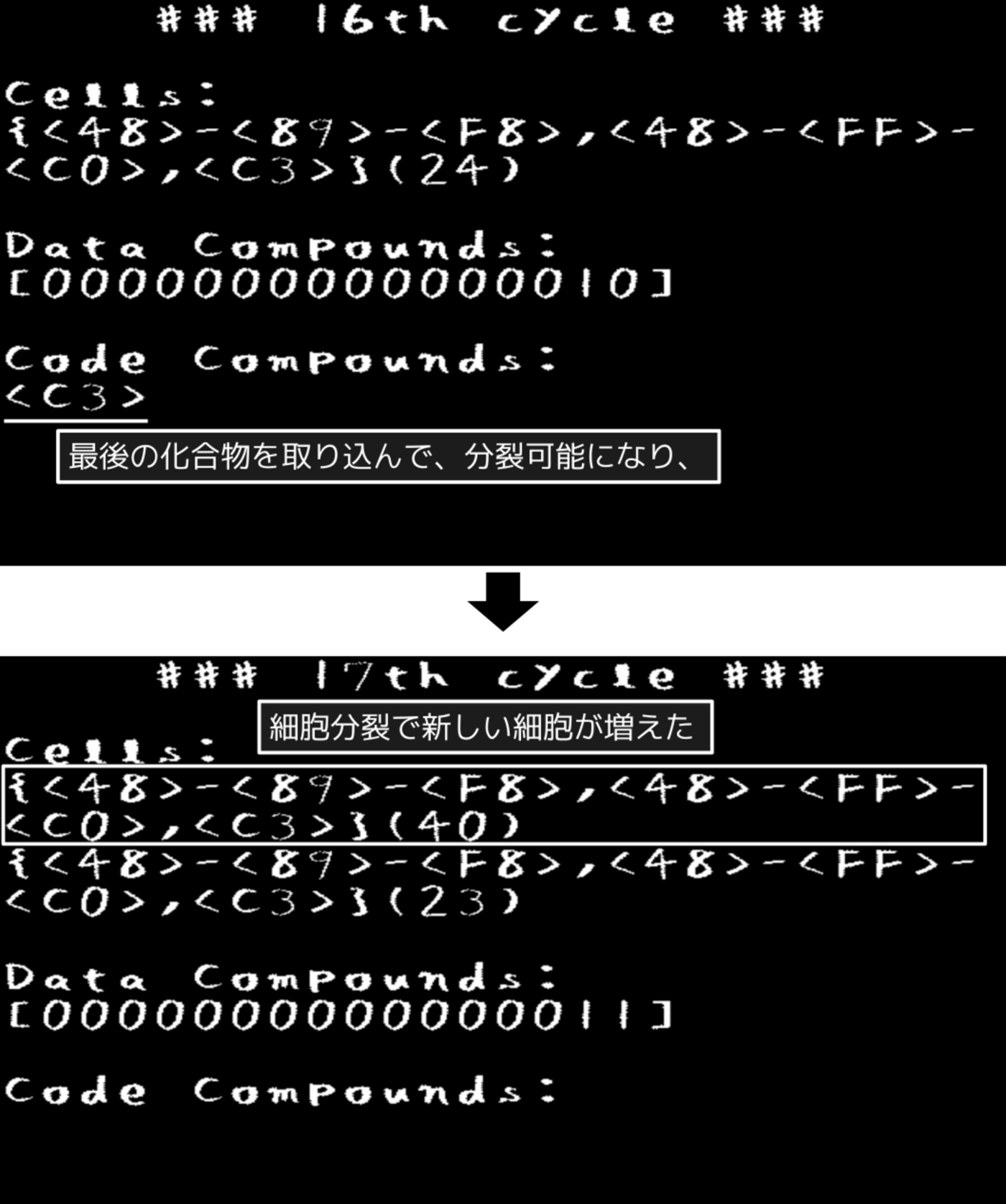
図3.4: PoCプログラムの実行画面(周期16 - 17)
細胞分裂すると、インクリメンタ細胞が2つになるため、データ化合物は2ずつ増加するようになります(図3.5)。
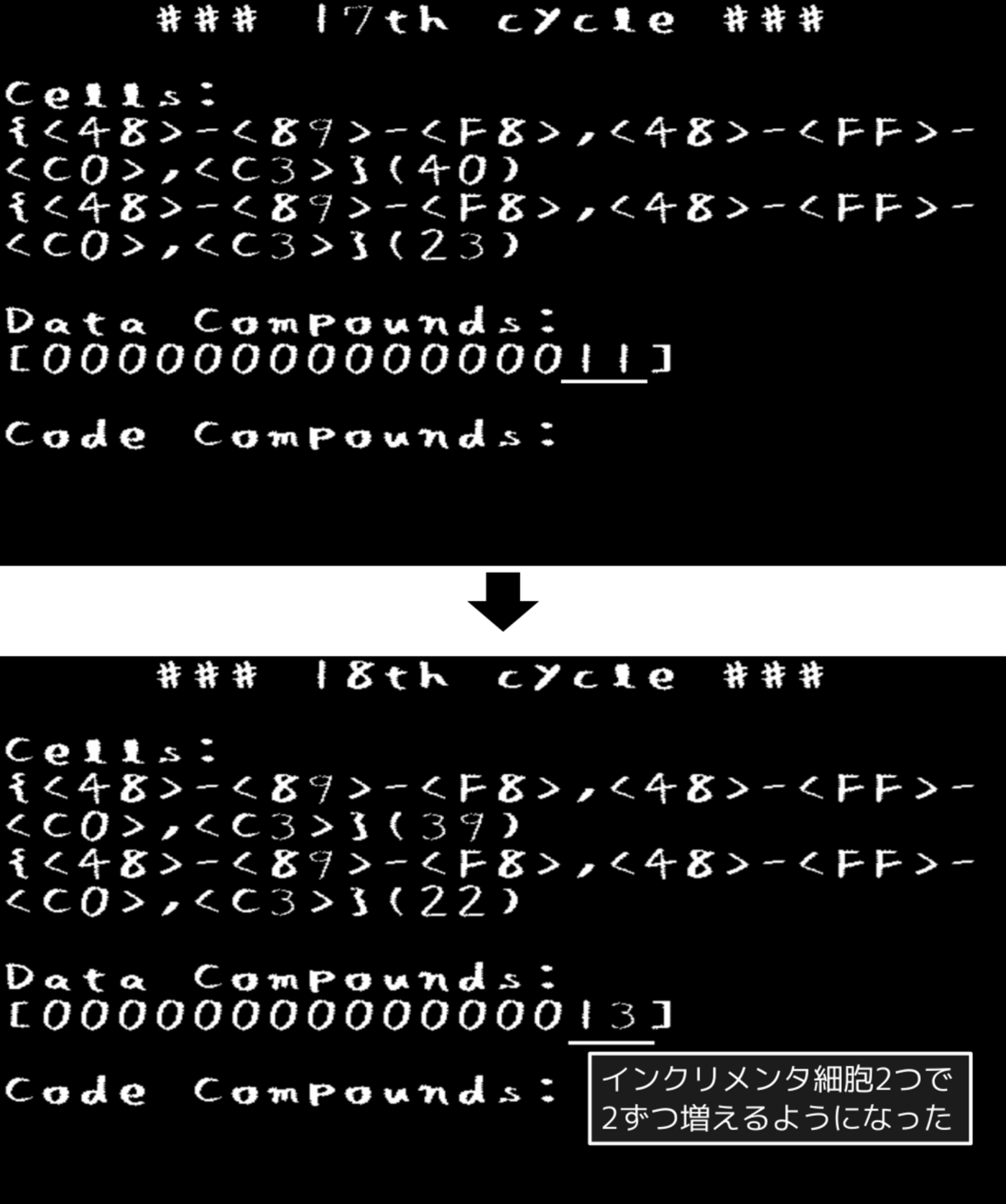
図3.5: PoCプログラムの実行画面(周期17 - 18)
実験として、30周期目に片方の細胞がウイルスに感染するように処理系へ仕掛けを入れてみました。30周期目に若い方の細胞の2つ目のタンパク質(inc命令)の一番うしろの化合物を「0xc0」から「0xc8」へ変更してみました(図3.6)。
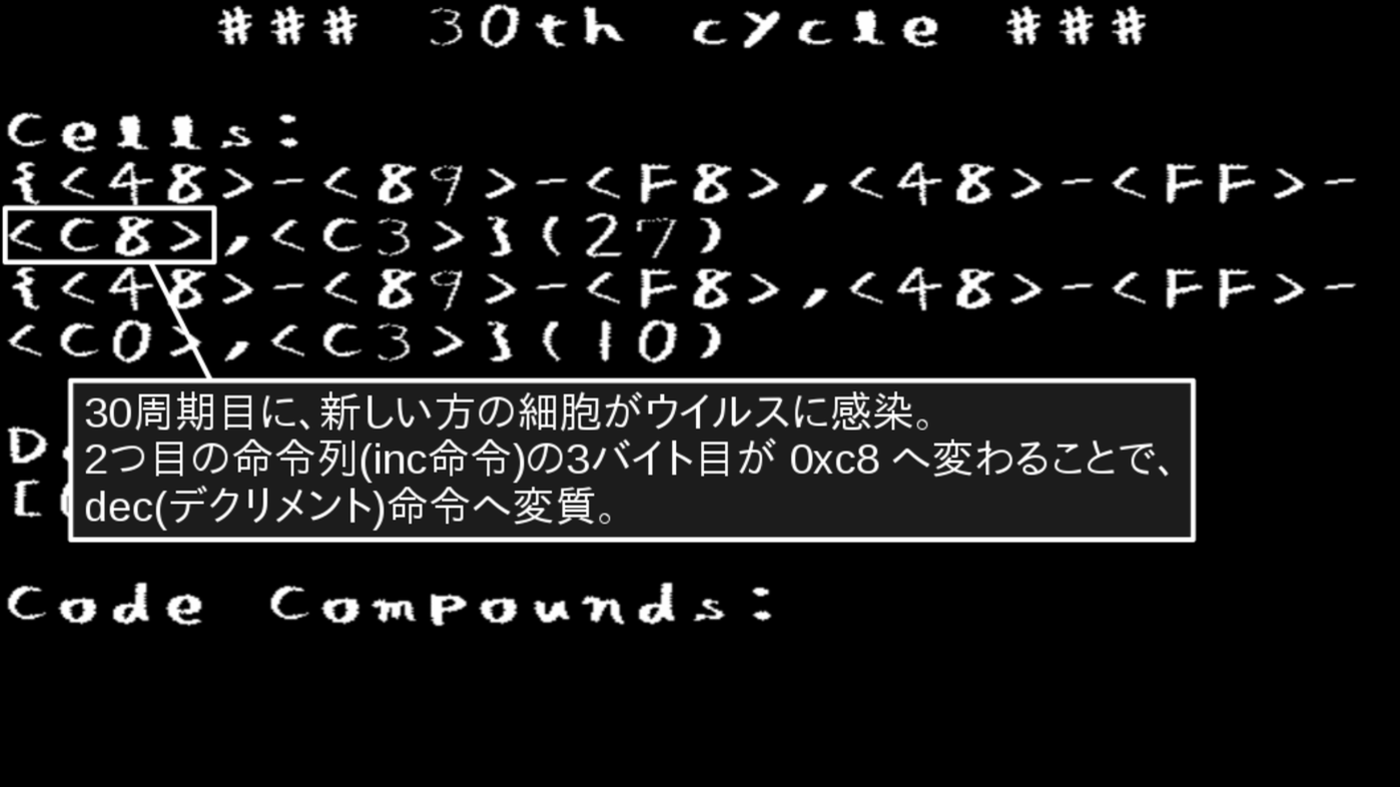
図3.6: PoCプログラムの実行画面(周期30)
宣伝: 「作って分かる!x86_64機械語入門」
図を順に紹介していく都合上、余白ができてしまったので宣伝を。
本書をもし試す際は機械語の知識があると良いです。当サークルの既刊で、一つ一つ命令や構文を理解してくことで簡単なプログラムを作る機械語の入門本を出しています(図3.7)。こちらも筆者のウェブサイト(「はじめに」参照)で公開していますので興味があれば見てみてください。

図3.7: 作って分かる!x86_64機械語入門
inc命令の最後のバイトが「0xc8」になっていると、「デクリメント(dec)命令」となるため、片方の細胞のインクリメントと相殺してデータ化合物が増減しなくなります(図3.8)。これは、インクリメンタのみの世界観であるはずのこの環境としては異常な状態(異物あるいは異常者が存在している状態)です。
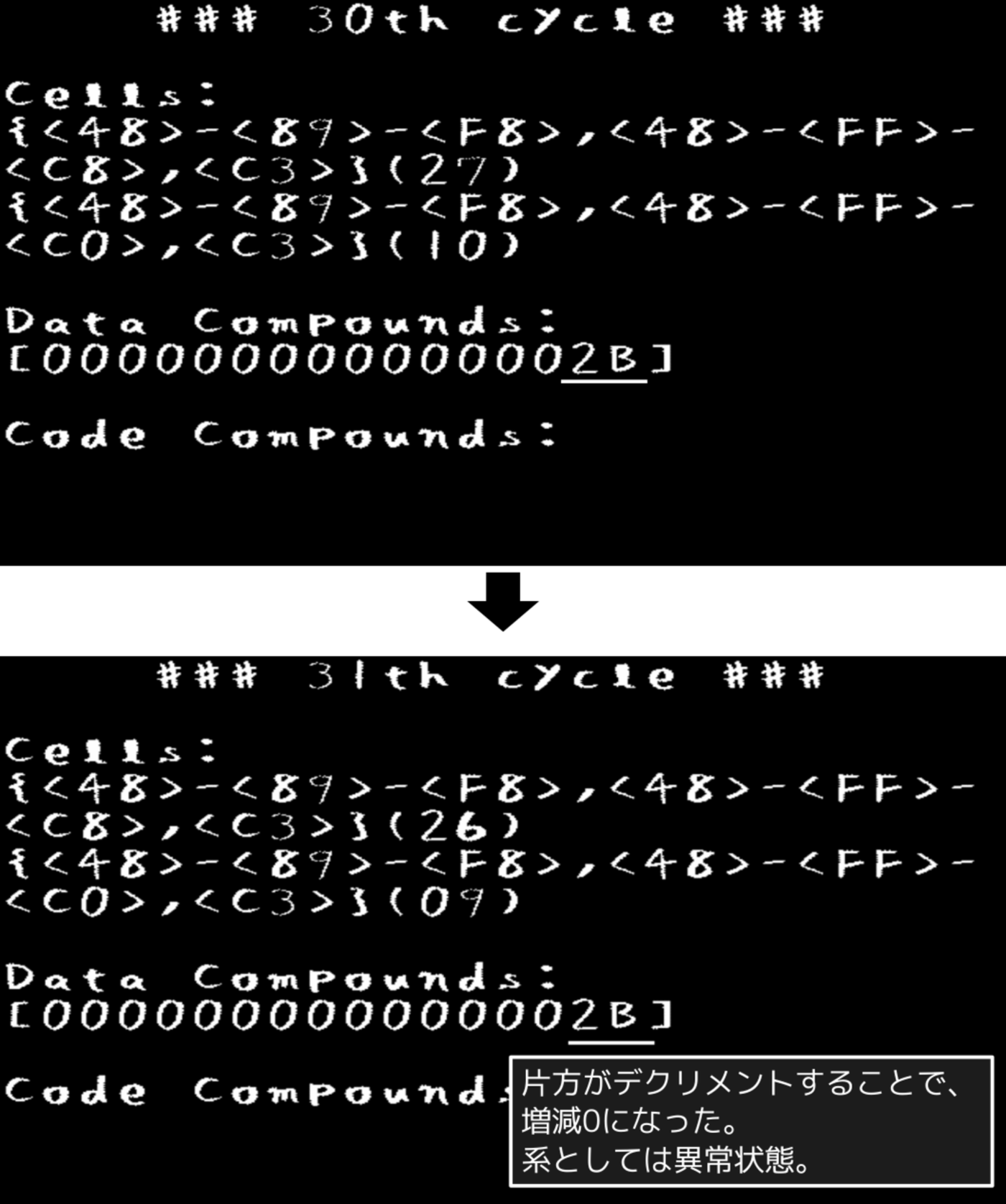
図3.8: PoCプログラムの実行画面(周期30 - 31)
そこから進むと、古い細胞(ウイルス感染していない方)が寿命を迎え、死滅します。細胞は死ぬ際、自身を構成する要素を化合物のレベルまで分解して環境へ放出します(図3.9)。
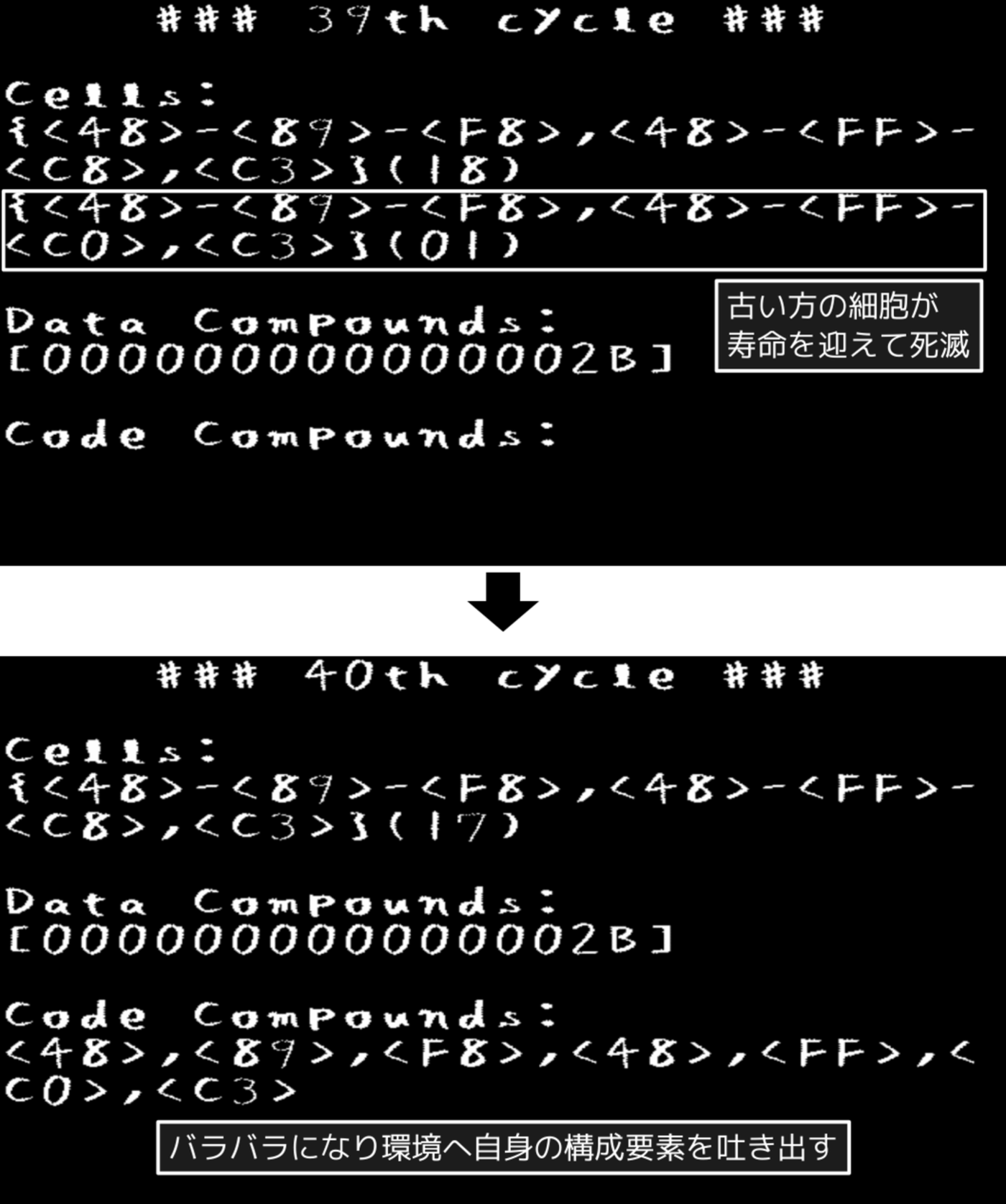
図3.9: PoCプログラムの実行画面(周期39 - 40)
すると、先ほどと同様に、環境に放出された化合物を現在生きている細胞が取り込んでいきます。デクリメンタへ変質してしまっているので、その間、データ化合物はデクリメントしていきます。ただし、DNAのコドンは変質していないため、inc命令の「0xc0」も取り込みます(DNAは表示させていないため画面上は変質していない事が分からないですが)(図3.10)。
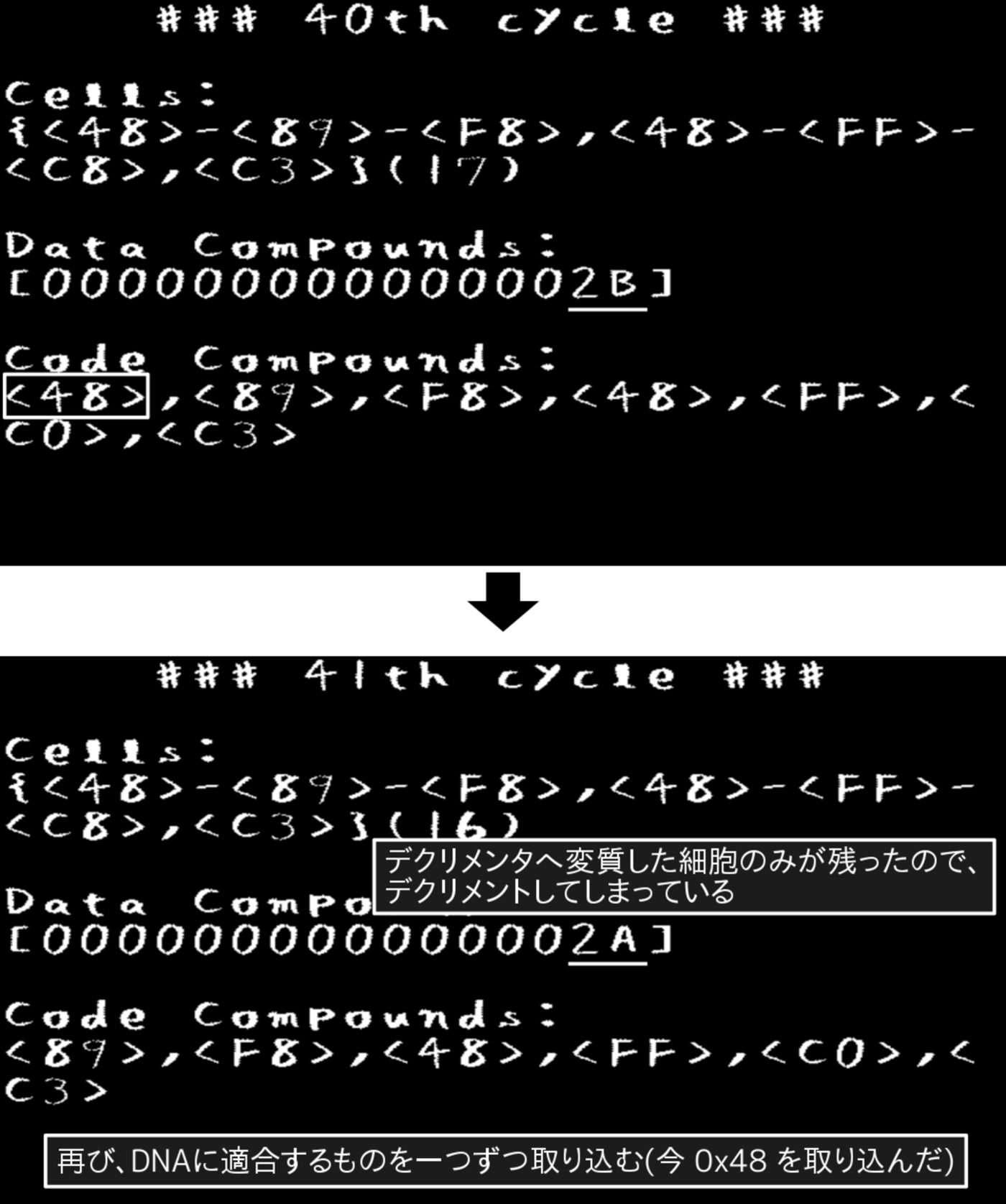
図3.10: PoCプログラムの実行画面(周期40 - 41)
全ての化合物を取り込むと、DNAに記載された化合物が全て揃うため分裂可能となり、細胞分裂を起こします。その際、先ほども説明した通り、DNAは変質していないため、分裂して生まれた子は正常なインクリメンタ細胞です(図3.11)。
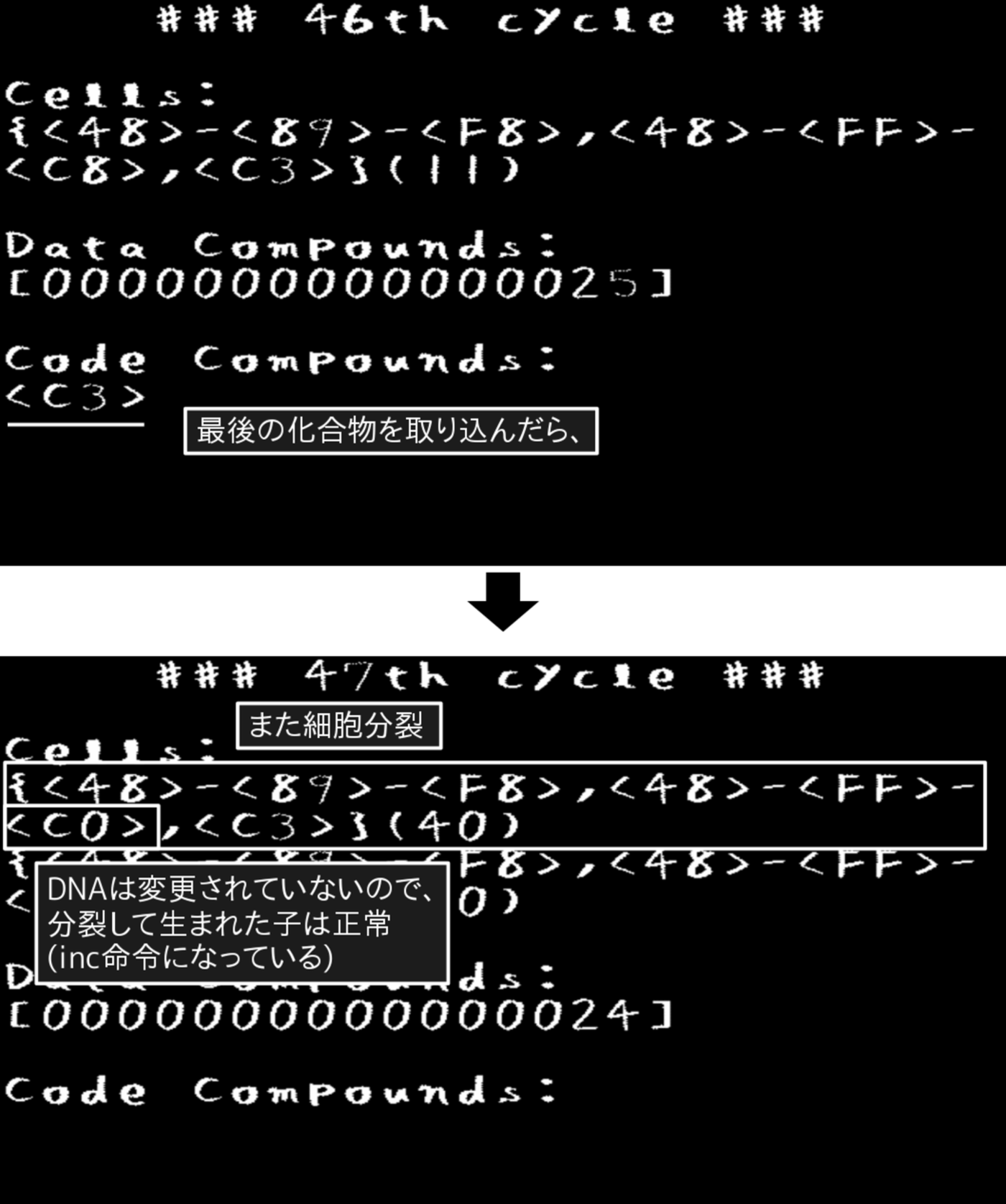
図3.11: PoCプログラムの実行画面(周期46 - 47)
そして、今度はウイルスに感染している方が寿命を迎えます。細胞を構成するinc命令の化合物が一箇所「0xc8」に変質しているため、それが環境へ放出されています(図3.12)。
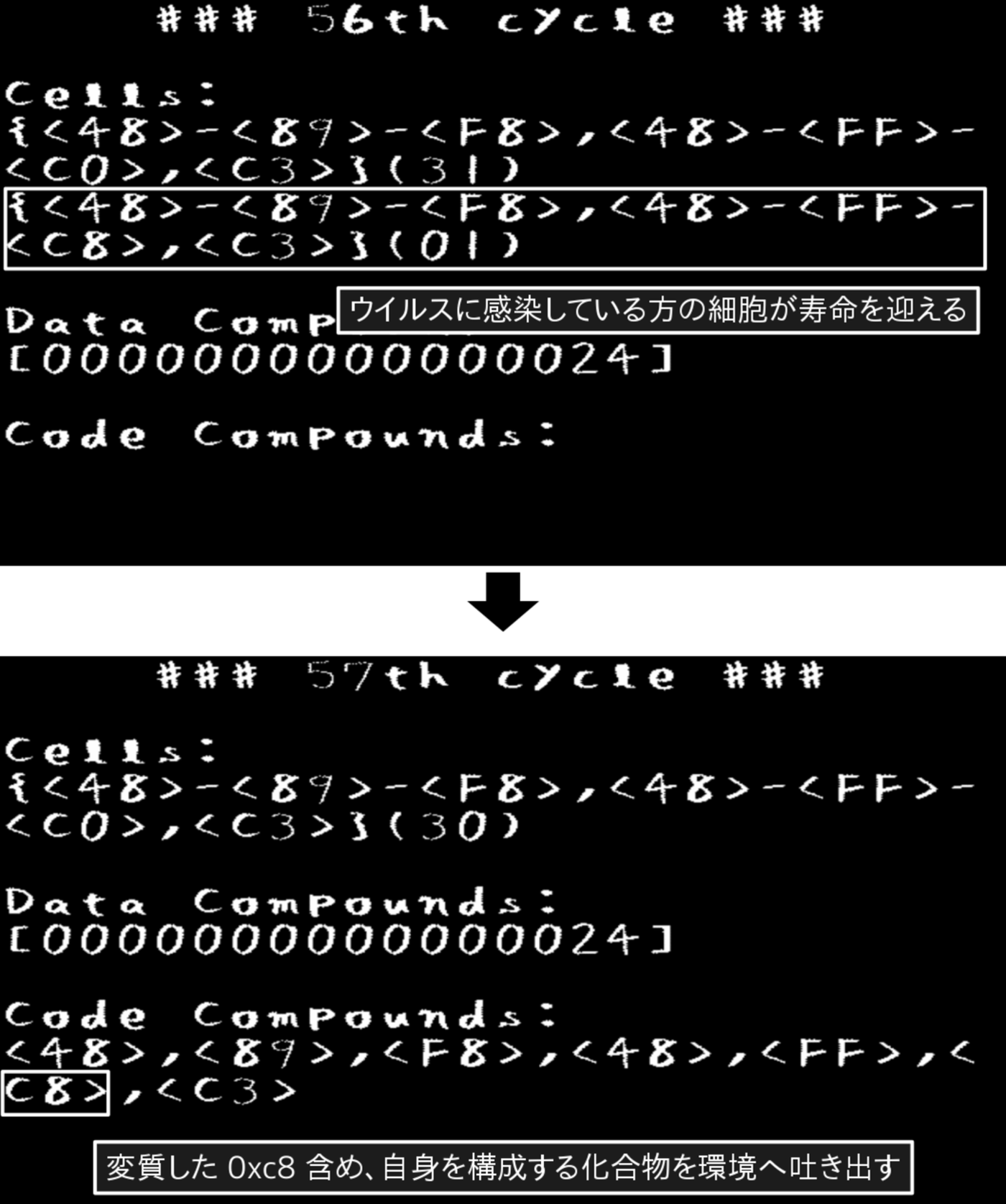
図3.12: PoCプログラムの実行画面(周期56 - 57)
すると、再び残された細胞が放出された化合物を取り込んでいきます(図3.13)。
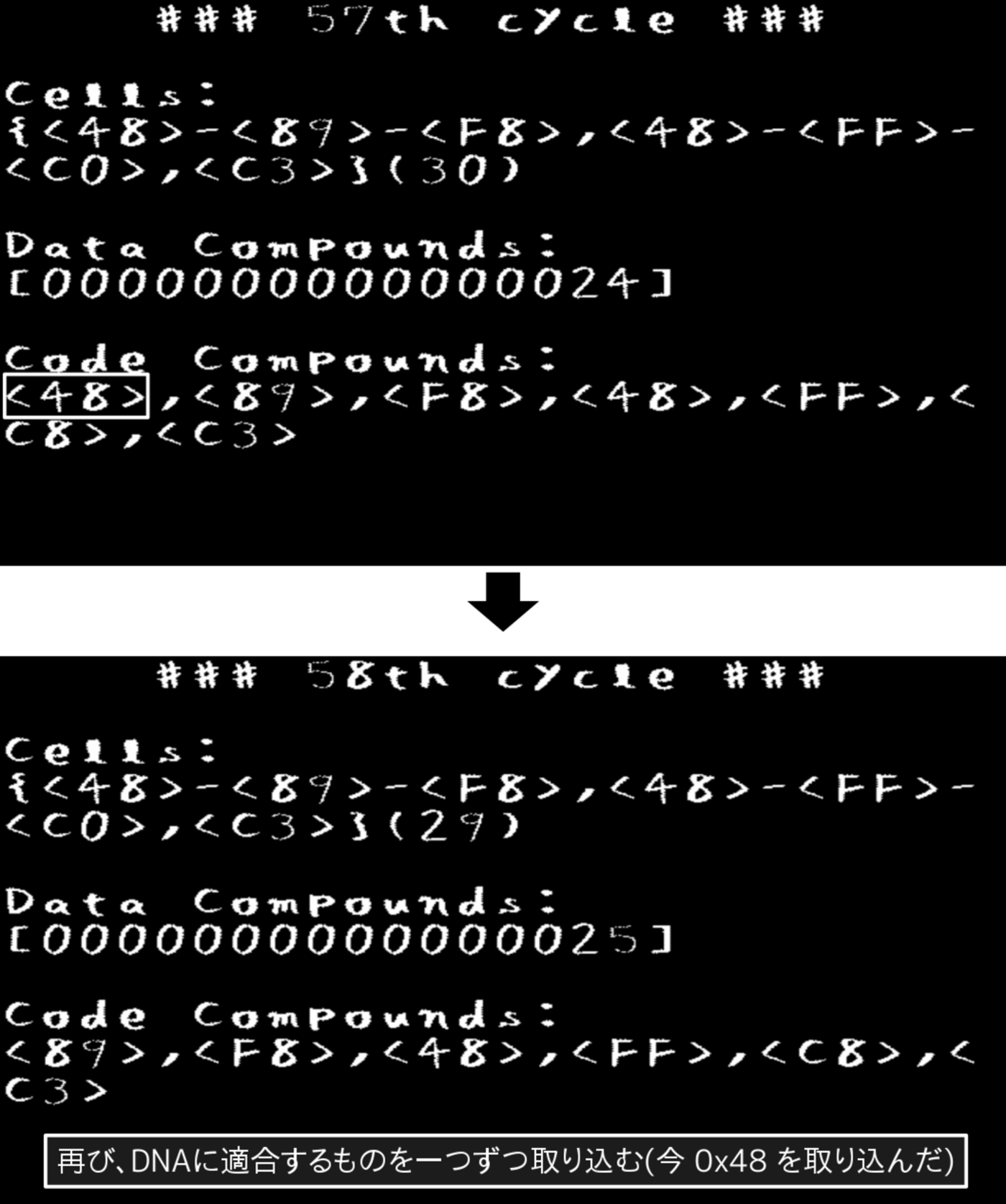
図3.13: PoCプログラムの実行画面(周期57 - 58)
ただし、変質した「0xc8」の化合物だけは正常なインクリメンタ細胞のDNAには無いため、取り込まれずに残ります(図3.14)。「デクリメンタが存在する」という異常状態は、「細胞はいずれ死ぬ」という機能により、異常状態から回復する事ができました。
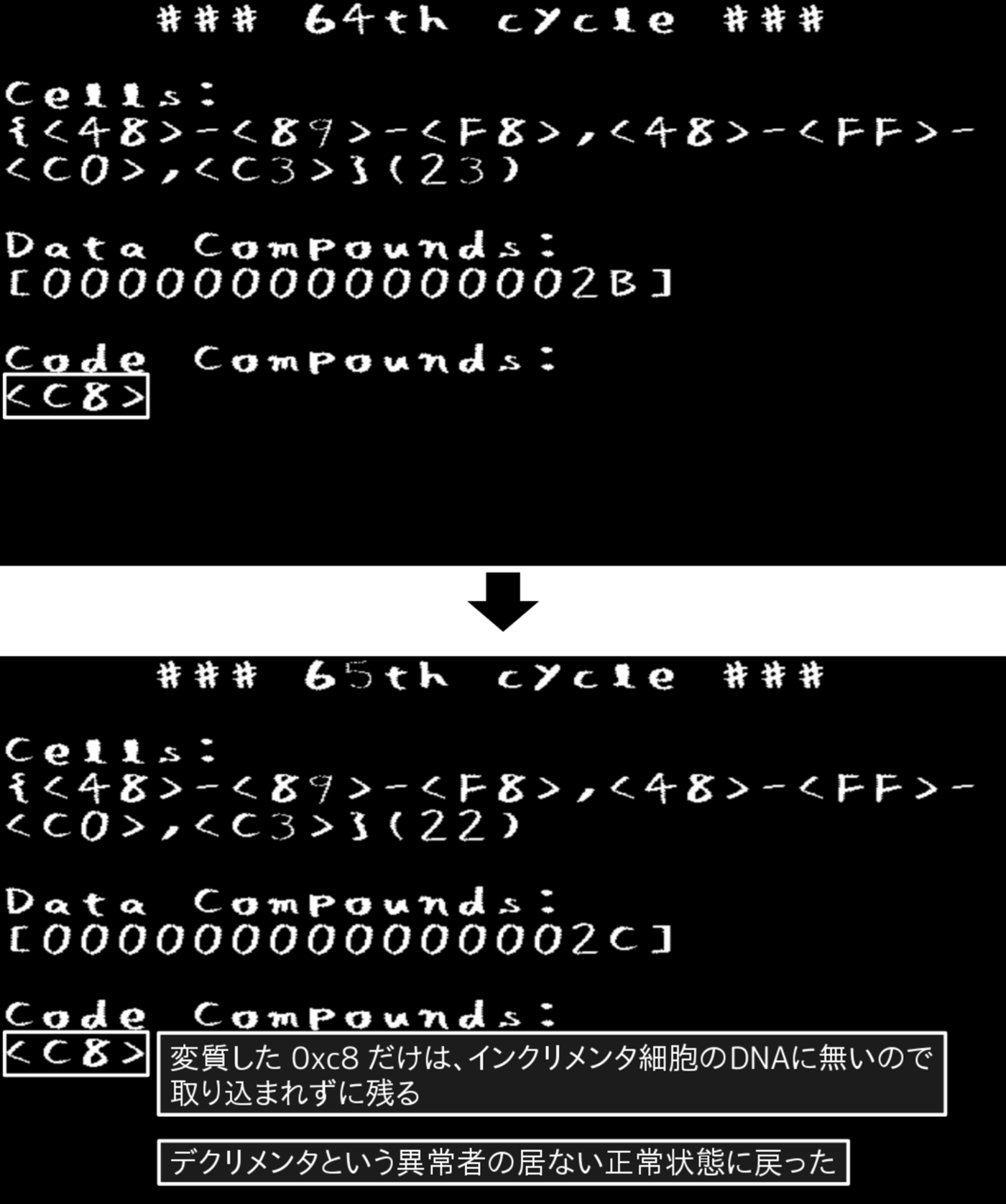
図3.14: PoCプログラムの実行画面(周期64 - 65)
なお、inc命令を構成する化合物である「0xc0」が足りないため、残った細胞は細胞分裂を行うことができず、寿命を迎えて死滅します。このインクリメンタ細胞が繁栄してから滅亡するまでの結果、データ化合物に最終的に記録された数字は「0x42」でした(図3.15)。
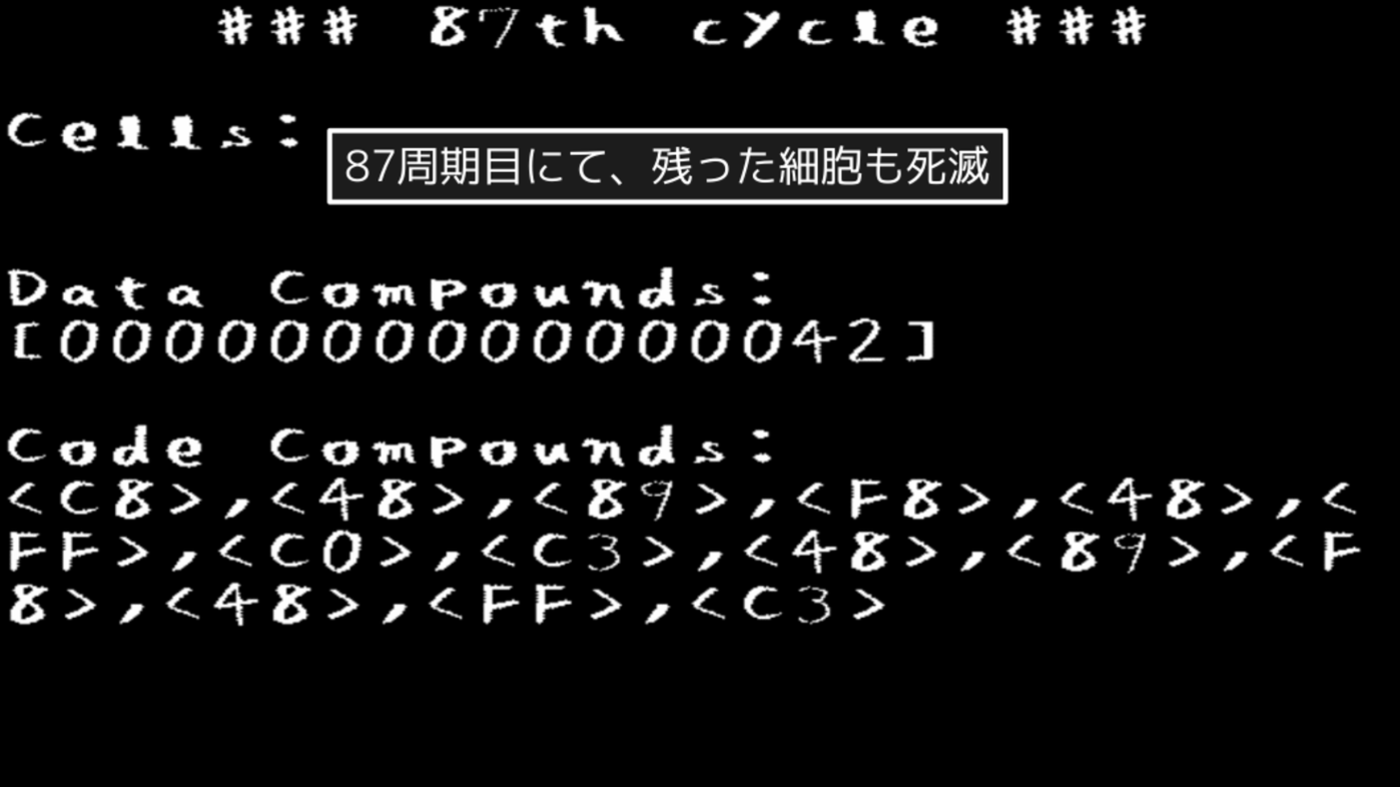
図3.15: PoCプログラムの実行画面(周期87)
3.3 まとめ
この章では、前章で説明した単細胞生物と処理系の実装について特筆事項を紹介し、PoC実装の実行結果を紹介しました。
実行結果から、本書で目的としている「生きていると言い張る」ための「5つの振る舞い」が実際に動作していることを確認できました。